針對二維電泳凝膠圖像匹配過程中,由于圖像局部非線性形變導致的偽匹配和漏匹配問題,本文提出了一種結合灰度分層和幾何分塊的自動匹配算法。首先,依據灰度和幾何位置對蛋白質點進行分組,采用形狀上下文特征,結合歸一化互相關法對蛋白質點進行粗匹配;然后,以粗匹配結果作為標記特征點,采用幾何相似性準則,對未匹配點進行精確匹配;最后,采用局部仿射變換模型,驗證匹配的正確性,去除偽匹配和漏匹配。通過對不同來源的凝膠圖像進行匹配實驗,結果表明本算法能有效解決蛋白質點匹配中的偽匹配和漏匹配問題,可獲得更為精確的匹配結果。
引用本文: 唐浩, 熊邦書, 歐巧鳳, 李俊. 基于灰度分層和幾何分塊的蛋白質點匹配算法. 生物醫學工程學雜志, 2014, 31(3): 487-492,498. doi: 10.7507/1001-5515.20140090 復制
版權信息: ?四川大學華西醫院華西期刊社《生物醫學工程學雜志》版權所有,未經授權不得轉載、改編
引言
二維電泳(two-dimension electrophoresis,2-DE) 凝膠技術[1-2]是生物學研究中常用的蛋白質復合物分離技術,通過對二維電泳凝膠圖像進行蛋白質點檢測、匹配和描述等操作,提取差異蛋白質點,為疾病診斷、藥物研制和環境污染分析提供依據。蛋白質點匹配是差異蛋白質點提取的關鍵環節,是從不同二維電泳凝膠圖像中找出同源蛋白質點的過程。由于不同凝膠圖像間存在差異,如幾何形變、野點和偽蛋白質點干擾等,都會影響匹配精度,所以需選擇合適的描述特征來衡量蛋白質點間的相似度。常用的蛋白質點特征描述方法有基于灰度[3-4]和基于幾何[5]兩種。Al-Tam等[6]采用蛋白質點灰度、體積和相關鄰近圖 (relative neighborhood graph,RNG)結構作為描述特征,能快速得出匹配結果,但RNG的結構易受偽蛋白質點和野點影響。熊邦書等[7]采用蛋白質點中心灰度、局部重疊率以及蛋白質點間構成的向量集作為描述特征,結合分層策略進行匹配,能夠匹配大多數同源蛋白質點,但在處理灰度特征相似的蛋白質點時易出現偽匹配。在匹配過程中,選取合適的變換模型校正圖像形變也是匹配成功的關鍵。常用的變換模型有線性[8]和非線性[9]兩類。Horaud等[10]提出一種基于剛性變換疊加的變換模型,并采用最大似然估計方法訓練剛性變換參數,該模型可以描述剛性和非剛性形變,但對幾何位置相近的點存在匹配誤差。Rogers等[11]采用了彈性變換模型,能很好地描述局部形變,但算法復雜度較高。
本文在文獻[7]的基礎上,采用幾何分塊和灰度分層相結合的匹配策略,結合形狀上下文(shape context)特征[12]與歸一化互相關法對蛋白質點進行粗匹配,并在精確匹配之后建立局部仿射變換模型,檢查匹配正確性,去除偽匹配和漏匹配,提高了匹配的精度。
1 算法描述
1.1 蛋白質點粗匹配
1.1.1 蛋白質點分層
在凝膠圖像中,每個蛋白質點都有不同的灰度特征,為減少無關蛋白質點間匹配造成的冗余運算,本文采用灰度分層法對檢測到的蛋白質點按中心灰度值進行分層,對同一灰度層級的蛋白質點進行匹配。對于參考圖像Ia和目標圖像Ib,其灰度分層的步長Sa和Sb分別按式(1)和式(2)計算,即
| ${{S}_{a}}=({{l}_{amax}}-{{l}_{amin}})/3,$ |
| ${{S}_{b}}=({{l}_{bmax}}-{{l}_{bmin}})/3,$ |
式中lamax和lamin分別為參考圖像Ia中蛋白質點的灰度最大和最小值,lbmax和lbmin分別為目標圖像Ib中蛋白質點的灰度最大和最小值。
為減少圖像亮度和對比度差異對分層結果的影響,需對參考和目標圖像進行歸一化,將它們的灰度分布整合到同一區間內。
1.1.2 蛋白質點分塊
為了進一步提高蛋白質點的匹配速度和精度,在蛋白質點分層之后,將整幅凝膠圖像劃分為R行和C列的子區域,R和C分別按式(3)和式(4)計算,即
| $R=[H/(5DT)],$ |
| $C=[W/(5DT)],$ |
式中H和W分別為圖像的寬和高,DT為圖像中心寬W/2、高H/2區域內兩兩最鄰近點間的距離均值。
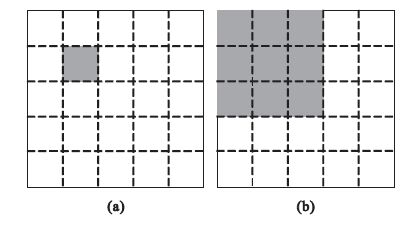
將蛋白質點按坐標劃分至各區塊中,各蛋白點的匹配搜索區域由整幅圖像縮小為相同及鄰近區塊,如圖 1所示,可減少不同區塊間無關蛋白質點的匹配,同時將復雜的幾何畸變化整為零,可提高匹配速度。
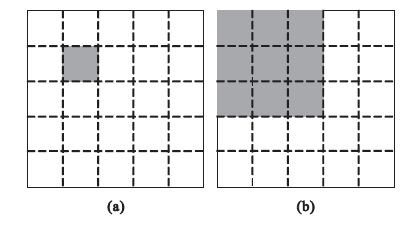 圖1
蛋白質點分塊匹配策略示意圖
圖1
蛋白質點分塊匹配策略示意圖
(a)參考圖像;(b)目標圖像
Figure1. Diagram of geometric blocking method(a) reference image; (b) target image
1.1.3 形狀描述和歸一化互相關結合的粗匹配
為提高蛋白質點粗匹配精度,在文獻[7]的基礎上,本文增加了幾何特征作為相關性判定條件,采用形狀上下文和歸一化互相關相結合的相似性度量方法完成蛋白質點粗匹配。
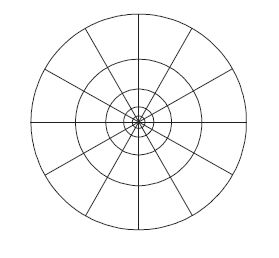
蛋白質點的形狀上下文特征體現了其鄰近點數量和分布情況,由形狀直方圖表示。設凝膠圖像中共有n個蛋白質點,要計算其中Pi點的特征,首先需將Pi作為坐標原點,計算剩余n-1個點的對數極坐標;然后,以Pi為圓心、R為半徑,按對數距離間隔建立M個同心圓,并沿圓周方向N等分,形成包含M×N個子塊的靶狀模板,如圖 2所示;最后,計算剩余n-1個點與Pi的位置關系,并將其劃分至模板中的對應子區塊內。統計落入各子區塊的蛋白質點數量,可得出點Pi的形狀直方圖。
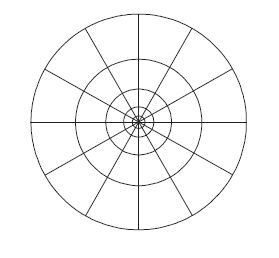 圖2
Shape Context模板
Figure2.
Template of Shape Context
圖2
Shape Context模板
Figure2.
Template of Shape Context
通過計算形狀直方圖間的χ2距離可確定蛋白質點的相似性。點pai與點pbj的相似性按式(5)計算,即
| $S{{c}_{ai,bj}}=\frac{1}{2}\sum\limits_{k=1}^{K}{\frac{{{[{{h}_{i}}\left( k \right)-{{h}_{j}}\left( k \right)]}^{2}}}{{{h}_{i}}\left( k \right)+{{h}_{j}}\left( k \right)}}$ |
式中hi(k)和hj(k)分別為點pai和點pbj的歸一化形狀直方圖,k為靶狀模板的子區塊編號,K=M×N。在hi(k)和hj(k)不為0時,落在相同子區塊中的近鄰點數目越相近,Scai,bj兩點相似度越大,反之越小,且0≤Scai,bj≤1。
本文粗匹配相關性度量是由形狀上下文相似度Scai,bj、灰度相似性度量Rai,bj和圖像重疊率Oai,bj[7]決定。對于圖像Ia和圖像Ib中兩個待匹配蛋白質點pai和點pbj,兩點的相關性度量值Dai,bj按式(6)計算,即
| ${{D}_{ai,bj}}=\alpha (1-S{{c}_{ai,bj}})+\beta ~{{R}_{ai,bj}}+\gamma ~{{O}_{ai,bj}},$ |
式中α、β和γ為權重參數,且α+β+γ=1。Dai,bj越大,兩點越相似。對于點pai,若點pbj與其的相似度最大且滿足Dai,bj≥0.7,則判斷蛋白質點pai和點pbj為相關點對。
最后,結合分層和分塊策略,對同一灰度層級內各區塊的蛋白質點分別進行相關性度量,保存相關點對,完成粗匹配。
1.2 蛋白質點精確匹配
1.2.1 基于幾何相關的整體匹配
針對粗匹配后剩余的未匹配點,本文將粗匹配結果作為標記特征點,用多項式擬合方法確定兩圖像間的整體幾何變換關系。對于待匹配點,采用其鄰近已匹配點間的有序矢量集計算相似度,從而獲得凝膠圖像的整體匹配結果[7]。
1.2.2 基于仿射變換的局部檢驗
經整體匹配后,凝膠圖像中大部分同源蛋白質點都已匹配成功。為保證匹配精度,本文利用蛋白質點鄰近已匹配點集建立局部坐標變換模型的方法,對匹配結果去偽查漏。
局部變換模型采用仿射變換,其變換方程如式(7)所示,即
| $\left[ \matrix{ {a_1}{a_2}{a_3} \hfill \cr {b_1}{b_2}{b_3} \hfill \cr} \right]\left( \matrix{ x \hfill \cr y \hfill \cr 1 \hfill \cr} \right) = \left( \matrix{ x\prime \hfill \cr y\prime \hfill \cr} \right)$ |
式中點(x′,y′)為點(x,y)的變換坐標,(a1a2a3)和(b1b2b3)為仿射變換系數,可由式(8)和式(9)求出,即
| $\left( \matrix{ \Sigma {x_a}\Sigma {y_a}n \hfill \cr \Sigma {x_a}^2\Sigma {x_a}{y_a}\Sigma {x_a} \hfill \cr \Sigma {x_a}{y_a}\Sigma {y_a}^2\Sigma {y_a} \hfill \cr} \right)\cdot\left( \matrix{ {a_1} \hfill \cr {a_2} \hfill \cr {a_3} \hfill \cr} \right) = \left( \matrix{ \Sigma {x_b} \hfill \cr \Sigma {x_a}{x_b} \hfill \cr \Sigma {y_a}{x_b} \hfill \cr} \right),$ |
| $\left( \matrix{ \Sigma {x_a}\Sigma {y_a}n \hfill \cr \Sigma {x_a}^2\Sigma {x_a}{y_a}\Sigma {x_a} \hfill \cr \Sigma {x_a}{y_a}\Sigma {y_a}^2\Sigma {y_a} \hfill \cr} \right)\cdot\left( \matrix{ {a_1} \hfill \cr {a_2} \hfill \cr {a_3} \hfill \cr} \right) = \left( \matrix{ \Sigma {y_b} \hfill \cr \Sigma {x_a}{x_b} \hfill \cr \Sigma {y_a}{y_b} \hfill \cr} \right),$ |
式中點(xa,ya)和點(xb,yb)為一對理論匹配點,n為理論點對數。要正確計算仿射變換系數,須保證n≥3。
假設參考圖像Ia中點pas與目標圖像Ib中點pbt為一對匹配點,要驗證它們的匹配正確性,需選取點pas鄰域內的已匹配點作為理論匹配點,計算仿射變換系數。鄰域半徑Ras按式(10)計算,即
| $Ras = {{\sqrt 2 } \over 2}max\left( {w,h} \right),$ |
式中w、h分別表示凝膠圖像分塊子區域的寬和高。
計算點pas在目標圖像Ib中的仿射變換映射點p′as。若在圖像Ib中點pbt距點p′as最近,則匹配正確,否則為偽匹配。若匹配正確,將點pbt與點p′as的歐式距離作為點pas的匹配距離。
偽匹配檢驗之后,需檢測是否存在漏匹配。設點pau為參考圖像Ia中的未匹配蛋白質點,需建立局部仿射變換模型,計算點pau在目標圖像Ib中的映射點p′au,并將點pau鄰域內已匹配點中的最大匹配距離作為距離閾值Dt。
計算目標圖像Ib中的未匹配點與點p′au的歐氏距離,選擇距離小于Dt的點為候選匹配點。若點pbv為點pau的候選匹配點且距點p′au最近,則點pbv為點pau的相關點,建立匹配關系。
2 實驗及結果分析
為了驗證算法的有效性,在Windows XP環境下,利用C++ Builder 6.0對算法進行編程實現。本文采用了亮度差異、幾何形變差異、欠飽和與過飽和三組圖像進行匹配實驗,并與文獻[7]的方法進行對比。
2.1 匹配誤差計算
為量化評估算法的匹配性能,本文采用式(11)和式(12)計算匹配誤差,即
| $\delta = {{{n_p} - {n_r}} \over {{n_p}}} \times 100\% ,$ |
| $f = {{{n_f}} \over {{n_r}}} \times 100\% ,$ |
式中δ為算法的漏匹配率,f為算法的偽匹配率,np為凝膠圖像中存在的同源蛋白質點對數,nr為算法獲得的正確相關點對數,nf為偽匹配點對數。
2.2 亮度差異圖像的匹配實驗
凝膠圖像拍攝過程中由于光源亮度的不同,易造成背景亮度不一致,給蛋白質點匹配造成困難。本組實驗選取了一對亮度不同的同源凝膠圖像進行實驗。結果如圖 3所示,標記“+”表示檢測到的蛋白質點中心位置,相同數字表示匹配到的蛋白質點點對。表 1為本文與文獻[7]算法的漏匹配率、偽匹配數和偽匹配率等量化對比結果。從表 1可以看出,本算法在圖像存在亮度差異時能保證良好的匹配精度。
 圖3
亮度差異圖組
圖3
亮度差異圖組
(a)參考圖像;(b)目標圖像
Figure3. Two images with different brightness(a) reference image; (b) target image
2.3 欠飽和與過飽和圖像的匹配實驗
不同的凝膠圖像由于成像參數不同,可能產生圖像欠飽和或過飽和。欠飽和圖像中蛋白質點與背景灰度差別較小,易出現蛋白質點漏檢;而過飽和圖像中由于一些蛋白質點沒有灰度尖峰,易導致灰度中心偏移。本組實驗分別選取了兩組圖像進行了匹配實驗,實驗結果分別如圖 4和圖 5所示,標記“+”表示檢測到的蛋白質點中心位置,相同數字表示匹配到的蛋白質點點對。表 2和表 3為本文與文獻[7]算法的漏匹配率、偽匹配數和偽匹配率等量化對比結果。從表 2和表 3可以看出,本文算法對欠飽和和過飽和圖像都有較好的匹配精度。
 圖4
欠飽和圖組
圖4
欠飽和圖組
(a)參考圖像;(b)目標圖像
Figure4. Undersaturation images(a) reference image; (b) target image
 圖5
過飽和圖組
圖5
過飽和圖組
(a)參考圖像;(b)目標圖像
Figure5. Supersaturation images(a) reference image; (b) target image
2.4 幾何形變差異圖像的匹配實驗
在制作凝膠電泳時,由于實驗中蛋白質存在電泳偏移率差異,易使圖像出現幾何形變。本組實驗選取了一組幾何形變不同的圖像進行了匹配實驗,實驗結果如圖 6所示,標記“+”表示檢測到的蛋白質點中心位置,相同數字表示匹配到的蛋白質點點對。表 4為本文與文獻[7]算法的的漏匹配率、偽匹配數和偽匹配率等量化對比結果。從表 4可以看出,本文算法對存在形變差異的圖像能保證良好的匹配精度。
 圖6
幾何形變差異圖組
圖6
幾何形變差異圖組
(a)參考圖像;(b)目標圖像
Figure6. Two images with different geometric distortion(a) reference image; (b) target image
3 結論
本文提出了基于灰度分層和幾何分塊的凝膠圖像同源蛋白質點匹配算法,算法在粗匹配中采用灰度分層和幾何分塊相結合的方法,并利用形狀上下文特征和歸一化互相關準則,實現部分特征明顯蛋白質點的匹配,克服了灰度相似非同源蛋白質點對粗匹配結果的影響,提高了匹配精度;在精確匹配中利用蛋白質點鄰近已匹配點的幾何相關度,實現大多數蛋白質點的匹配;在精確匹配后采用局部仿射變換模型,進行蛋白質點偽匹配和漏匹配檢測,進一步提高了匹配精度。通過多種不同類型的真實凝膠圖像匹配實驗,驗證了本文算法的有效性。
引言
二維電泳(two-dimension electrophoresis,2-DE) 凝膠技術[1-2]是生物學研究中常用的蛋白質復合物分離技術,通過對二維電泳凝膠圖像進行蛋白質點檢測、匹配和描述等操作,提取差異蛋白質點,為疾病診斷、藥物研制和環境污染分析提供依據。蛋白質點匹配是差異蛋白質點提取的關鍵環節,是從不同二維電泳凝膠圖像中找出同源蛋白質點的過程。由于不同凝膠圖像間存在差異,如幾何形變、野點和偽蛋白質點干擾等,都會影響匹配精度,所以需選擇合適的描述特征來衡量蛋白質點間的相似度。常用的蛋白質點特征描述方法有基于灰度[3-4]和基于幾何[5]兩種。Al-Tam等[6]采用蛋白質點灰度、體積和相關鄰近圖 (relative neighborhood graph,RNG)結構作為描述特征,能快速得出匹配結果,但RNG的結構易受偽蛋白質點和野點影響。熊邦書等[7]采用蛋白質點中心灰度、局部重疊率以及蛋白質點間構成的向量集作為描述特征,結合分層策略進行匹配,能夠匹配大多數同源蛋白質點,但在處理灰度特征相似的蛋白質點時易出現偽匹配。在匹配過程中,選取合適的變換模型校正圖像形變也是匹配成功的關鍵。常用的變換模型有線性[8]和非線性[9]兩類。Horaud等[10]提出一種基于剛性變換疊加的變換模型,并采用最大似然估計方法訓練剛性變換參數,該模型可以描述剛性和非剛性形變,但對幾何位置相近的點存在匹配誤差。Rogers等[11]采用了彈性變換模型,能很好地描述局部形變,但算法復雜度較高。
本文在文獻[7]的基礎上,采用幾何分塊和灰度分層相結合的匹配策略,結合形狀上下文(shape context)特征[12]與歸一化互相關法對蛋白質點進行粗匹配,并在精確匹配之后建立局部仿射變換模型,檢查匹配正確性,去除偽匹配和漏匹配,提高了匹配的精度。
1 算法描述
1.1 蛋白質點粗匹配
1.1.1 蛋白質點分層
在凝膠圖像中,每個蛋白質點都有不同的灰度特征,為減少無關蛋白質點間匹配造成的冗余運算,本文采用灰度分層法對檢測到的蛋白質點按中心灰度值進行分層,對同一灰度層級的蛋白質點進行匹配。對于參考圖像Ia和目標圖像Ib,其灰度分層的步長Sa和Sb分別按式(1)和式(2)計算,即
| ${{S}_{a}}=({{l}_{amax}}-{{l}_{amin}})/3,$ |
| ${{S}_{b}}=({{l}_{bmax}}-{{l}_{bmin}})/3,$ |
式中lamax和lamin分別為參考圖像Ia中蛋白質點的灰度最大和最小值,lbmax和lbmin分別為目標圖像Ib中蛋白質點的灰度最大和最小值。
為減少圖像亮度和對比度差異對分層結果的影響,需對參考和目標圖像進行歸一化,將它們的灰度分布整合到同一區間內。
1.1.2 蛋白質點分塊
為了進一步提高蛋白質點的匹配速度和精度,在蛋白質點分層之后,將整幅凝膠圖像劃分為R行和C列的子區域,R和C分別按式(3)和式(4)計算,即
| $R=[H/(5DT)],$ |
| $C=[W/(5DT)],$ |
式中H和W分別為圖像的寬和高,DT為圖像中心寬W/2、高H/2區域內兩兩最鄰近點間的距離均值。
將蛋白質點按坐標劃分至各區塊中,各蛋白點的匹配搜索區域由整幅圖像縮小為相同及鄰近區塊,如圖 1所示,可減少不同區塊間無關蛋白質點的匹配,同時將復雜的幾何畸變化整為零,可提高匹配速度。
 圖1
蛋白質點分塊匹配策略示意圖
圖1
蛋白質點分塊匹配策略示意圖
(a)參考圖像;(b)目標圖像
Figure1. Diagram of geometric blocking method(a) reference image; (b) target image
1.1.3 形狀描述和歸一化互相關結合的粗匹配
為提高蛋白質點粗匹配精度,在文獻[7]的基礎上,本文增加了幾何特征作為相關性判定條件,采用形狀上下文和歸一化互相關相結合的相似性度量方法完成蛋白質點粗匹配。
蛋白質點的形狀上下文特征體現了其鄰近點數量和分布情況,由形狀直方圖表示。設凝膠圖像中共有n個蛋白質點,要計算其中Pi點的特征,首先需將Pi作為坐標原點,計算剩余n-1個點的對數極坐標;然后,以Pi為圓心、R為半徑,按對數距離間隔建立M個同心圓,并沿圓周方向N等分,形成包含M×N個子塊的靶狀模板,如圖 2所示;最后,計算剩余n-1個點與Pi的位置關系,并將其劃分至模板中的對應子區塊內。統計落入各子區塊的蛋白質點數量,可得出點Pi的形狀直方圖。
 圖2
Shape Context模板
Figure2.
Template of Shape Context
圖2
Shape Context模板
Figure2.
Template of Shape Context
通過計算形狀直方圖間的χ2距離可確定蛋白質點的相似性。點pai與點pbj的相似性按式(5)計算,即
| $S{{c}_{ai,bj}}=\frac{1}{2}\sum\limits_{k=1}^{K}{\frac{{{[{{h}_{i}}\left( k \right)-{{h}_{j}}\left( k \right)]}^{2}}}{{{h}_{i}}\left( k \right)+{{h}_{j}}\left( k \right)}}$ |
式中hi(k)和hj(k)分別為點pai和點pbj的歸一化形狀直方圖,k為靶狀模板的子區塊編號,K=M×N。在hi(k)和hj(k)不為0時,落在相同子區塊中的近鄰點數目越相近,Scai,bj兩點相似度越大,反之越小,且0≤Scai,bj≤1。
本文粗匹配相關性度量是由形狀上下文相似度Scai,bj、灰度相似性度量Rai,bj和圖像重疊率Oai,bj[7]決定。對于圖像Ia和圖像Ib中兩個待匹配蛋白質點pai和點pbj,兩點的相關性度量值Dai,bj按式(6)計算,即
| ${{D}_{ai,bj}}=\alpha (1-S{{c}_{ai,bj}})+\beta ~{{R}_{ai,bj}}+\gamma ~{{O}_{ai,bj}},$ |
式中α、β和γ為權重參數,且α+β+γ=1。Dai,bj越大,兩點越相似。對于點pai,若點pbj與其的相似度最大且滿足Dai,bj≥0.7,則判斷蛋白質點pai和點pbj為相關點對。
最后,結合分層和分塊策略,對同一灰度層級內各區塊的蛋白質點分別進行相關性度量,保存相關點對,完成粗匹配。
1.2 蛋白質點精確匹配
1.2.1 基于幾何相關的整體匹配
針對粗匹配后剩余的未匹配點,本文將粗匹配結果作為標記特征點,用多項式擬合方法確定兩圖像間的整體幾何變換關系。對于待匹配點,采用其鄰近已匹配點間的有序矢量集計算相似度,從而獲得凝膠圖像的整體匹配結果[7]。
1.2.2 基于仿射變換的局部檢驗
經整體匹配后,凝膠圖像中大部分同源蛋白質點都已匹配成功。為保證匹配精度,本文利用蛋白質點鄰近已匹配點集建立局部坐標變換模型的方法,對匹配結果去偽查漏。
局部變換模型采用仿射變換,其變換方程如式(7)所示,即
| $\left[ \matrix{ {a_1}{a_2}{a_3} \hfill \cr {b_1}{b_2}{b_3} \hfill \cr} \right]\left( \matrix{ x \hfill \cr y \hfill \cr 1 \hfill \cr} \right) = \left( \matrix{ x\prime \hfill \cr y\prime \hfill \cr} \right)$ |
式中點(x′,y′)為點(x,y)的變換坐標,(a1a2a3)和(b1b2b3)為仿射變換系數,可由式(8)和式(9)求出,即
| $\left( \matrix{ \Sigma {x_a}\Sigma {y_a}n \hfill \cr \Sigma {x_a}^2\Sigma {x_a}{y_a}\Sigma {x_a} \hfill \cr \Sigma {x_a}{y_a}\Sigma {y_a}^2\Sigma {y_a} \hfill \cr} \right)\cdot\left( \matrix{ {a_1} \hfill \cr {a_2} \hfill \cr {a_3} \hfill \cr} \right) = \left( \matrix{ \Sigma {x_b} \hfill \cr \Sigma {x_a}{x_b} \hfill \cr \Sigma {y_a}{x_b} \hfill \cr} \right),$ |
| $\left( \matrix{ \Sigma {x_a}\Sigma {y_a}n \hfill \cr \Sigma {x_a}^2\Sigma {x_a}{y_a}\Sigma {x_a} \hfill \cr \Sigma {x_a}{y_a}\Sigma {y_a}^2\Sigma {y_a} \hfill \cr} \right)\cdot\left( \matrix{ {a_1} \hfill \cr {a_2} \hfill \cr {a_3} \hfill \cr} \right) = \left( \matrix{ \Sigma {y_b} \hfill \cr \Sigma {x_a}{x_b} \hfill \cr \Sigma {y_a}{y_b} \hfill \cr} \right),$ |
式中點(xa,ya)和點(xb,yb)為一對理論匹配點,n為理論點對數。要正確計算仿射變換系數,須保證n≥3。
假設參考圖像Ia中點pas與目標圖像Ib中點pbt為一對匹配點,要驗證它們的匹配正確性,需選取點pas鄰域內的已匹配點作為理論匹配點,計算仿射變換系數。鄰域半徑Ras按式(10)計算,即
| $Ras = {{\sqrt 2 } \over 2}max\left( {w,h} \right),$ |
式中w、h分別表示凝膠圖像分塊子區域的寬和高。
計算點pas在目標圖像Ib中的仿射變換映射點p′as。若在圖像Ib中點pbt距點p′as最近,則匹配正確,否則為偽匹配。若匹配正確,將點pbt與點p′as的歐式距離作為點pas的匹配距離。
偽匹配檢驗之后,需檢測是否存在漏匹配。設點pau為參考圖像Ia中的未匹配蛋白質點,需建立局部仿射變換模型,計算點pau在目標圖像Ib中的映射點p′au,并將點pau鄰域內已匹配點中的最大匹配距離作為距離閾值Dt。
計算目標圖像Ib中的未匹配點與點p′au的歐氏距離,選擇距離小于Dt的點為候選匹配點。若點pbv為點pau的候選匹配點且距點p′au最近,則點pbv為點pau的相關點,建立匹配關系。
2 實驗及結果分析
為了驗證算法的有效性,在Windows XP環境下,利用C++ Builder 6.0對算法進行編程實現。本文采用了亮度差異、幾何形變差異、欠飽和與過飽和三組圖像進行匹配實驗,并與文獻[7]的方法進行對比。
2.1 匹配誤差計算
為量化評估算法的匹配性能,本文采用式(11)和式(12)計算匹配誤差,即
| $\delta = {{{n_p} - {n_r}} \over {{n_p}}} \times 100\% ,$ |
| $f = {{{n_f}} \over {{n_r}}} \times 100\% ,$ |
式中δ為算法的漏匹配率,f為算法的偽匹配率,np為凝膠圖像中存在的同源蛋白質點對數,nr為算法獲得的正確相關點對數,nf為偽匹配點對數。
2.2 亮度差異圖像的匹配實驗
凝膠圖像拍攝過程中由于光源亮度的不同,易造成背景亮度不一致,給蛋白質點匹配造成困難。本組實驗選取了一對亮度不同的同源凝膠圖像進行實驗。結果如圖 3所示,標記“+”表示檢測到的蛋白質點中心位置,相同數字表示匹配到的蛋白質點點對。表 1為本文與文獻[7]算法的漏匹配率、偽匹配數和偽匹配率等量化對比結果。從表 1可以看出,本算法在圖像存在亮度差異時能保證良好的匹配精度。
 圖3
亮度差異圖組
圖3
亮度差異圖組
(a)參考圖像;(b)目標圖像
Figure3. Two images with different brightness(a) reference image; (b) target image
2.3 欠飽和與過飽和圖像的匹配實驗
不同的凝膠圖像由于成像參數不同,可能產生圖像欠飽和或過飽和。欠飽和圖像中蛋白質點與背景灰度差別較小,易出現蛋白質點漏檢;而過飽和圖像中由于一些蛋白質點沒有灰度尖峰,易導致灰度中心偏移。本組實驗分別選取了兩組圖像進行了匹配實驗,實驗結果分別如圖 4和圖 5所示,標記“+”表示檢測到的蛋白質點中心位置,相同數字表示匹配到的蛋白質點點對。表 2和表 3為本文與文獻[7]算法的漏匹配率、偽匹配數和偽匹配率等量化對比結果。從表 2和表 3可以看出,本文算法對欠飽和和過飽和圖像都有較好的匹配精度。
 圖4
欠飽和圖組
圖4
欠飽和圖組
(a)參考圖像;(b)目標圖像
Figure4. Undersaturation images(a) reference image; (b) target image
 圖5
過飽和圖組
圖5
過飽和圖組
(a)參考圖像;(b)目標圖像
Figure5. Supersaturation images(a) reference image; (b) target image
2.4 幾何形變差異圖像的匹配實驗
在制作凝膠電泳時,由于實驗中蛋白質存在電泳偏移率差異,易使圖像出現幾何形變。本組實驗選取了一組幾何形變不同的圖像進行了匹配實驗,實驗結果如圖 6所示,標記“+”表示檢測到的蛋白質點中心位置,相同數字表示匹配到的蛋白質點點對。表 4為本文與文獻[7]算法的的漏匹配率、偽匹配數和偽匹配率等量化對比結果。從表 4可以看出,本文算法對存在形變差異的圖像能保證良好的匹配精度。
 圖6
幾何形變差異圖組
圖6
幾何形變差異圖組
(a)參考圖像;(b)目標圖像
Figure6. Two images with different geometric distortion(a) reference image; (b) target image
3 結論
本文提出了基于灰度分層和幾何分塊的凝膠圖像同源蛋白質點匹配算法,算法在粗匹配中采用灰度分層和幾何分塊相結合的方法,并利用形狀上下文特征和歸一化互相關準則,實現部分特征明顯蛋白質點的匹配,克服了灰度相似非同源蛋白質點對粗匹配結果的影響,提高了匹配精度;在精確匹配中利用蛋白質點鄰近已匹配點的幾何相關度,實現大多數蛋白質點的匹配;在精確匹配后采用局部仿射變換模型,進行蛋白質點偽匹配和漏匹配檢測,進一步提高了匹配精度。通過多種不同類型的真實凝膠圖像匹配實驗,驗證了本文算法的有效性。