藥物組合的協同作用能夠解決單一藥物療法的獲得耐藥性問題,對于癌癥等復雜疾病的治療具有巨大的潛力。在本項研究中,為了探索不同藥物分子間相互作用對于抗癌藥物療效的影響,我們提出了一種基于Transformer的深度學習預測模型——SMILESynergy。首先用藥物的文本數據——簡化分子線性輸入規范(SMILES)表征藥物分子,其次通過SMILES Enumeration生成藥物分子的異構體進行數據增強,然后利用Transformer中的注意力機制對數據增強后的藥物進行編解碼,最后連接一個多層感知器(MLP)獲得藥物的協同作用值。實驗結果表明我們的模型在O’Neil數據集的回歸分析中均方誤差為51.34,分類分析中準確率為0.97,預測性能均優于DeepSynergy和MulinputSynergy模型。SMILESynergy具有更好的預測性能,可輔助研究人員快速篩選最優藥物組合以提高癌癥治療效果。
引用本文: 張立強, 秦玉芳, 陳明. SMILESynergy:基于Transformer預訓練模型的抗癌藥物協同作用預測. 生物醫學工程學雜志, 2023, 40(3): 544-551. doi: 10.7507/1001-5515.202209043 復制
版權信息: ?四川大學華西醫院華西期刊社《生物醫學工程學雜志》版權所有,未經授權不得轉載、改編
0 引言
癌癥是一種高度異質性的疾病,不同類型的癌癥具有不同的遺傳變異和表觀遺傳變異[1],導致不同個體對藥物的反應和耐藥性[2]也不同,因此選擇最優的藥物組合來提高治療效果是癌癥治療的一個重要挑戰。傳統的化療藥物研究方法需要通過大量的臨床試驗進行藥物篩選和劑量確定,耗費時間和資源,并且可能使患者面臨不必要甚至有害的治療[3]。而且由于癌癥的異質性和個體差異性,不同患者對同一種藥物的反應和副作用也不同,使得制定治療方案更加困難。因此利用計算模型來探索高通量篩選[4]實驗產生的藥物組合數據具有重要意義。
近年來研究人員運用深度學習在抗癌藥物協同作用預測的研究中做了大量的工作。如Preuer等[5]提出的DeepSynergy模型,以O’Neil數據集[6]上的藥物化合物以及細胞系基因表達信息作為輸入,連接一個全連接前饋神經網絡(fully forward neural network,FFNN),得到了不錯的抗癌藥物組合協同作用預測結果。在藥物化合物以及細胞系基因表達信息的基礎上,Zhang等[7]加入細胞系的拷貝數和基因突變數據作為輸入,并提出了基于自編碼器(auto encoder)的深度學習模型AuDNNsynergy來預測具有協同作用的藥物組合。在文獻[7]輸入數據的基礎上,陳希等[8]通過一維卷積層對輸入數據進行降維預處理,進而提出了MulinputSynergy藥物預測模型,并在模型中加入殘差前饋神經層,最終提升了藥物組合預測效果。Sun等[9]在O’Neil數據集的藥物數據上引入張量分解方法進行預處理,然后將分解的結果作為特征用于訓練深度神經網絡(deep neural network,DNN)模型以預測藥物對的協同效應。
鑒于近年來深度學習在自然語言處理(natural language process,NLP)領域的發展,以Transformer[10]為代表的預訓練語言模型加下游任務的方案在藥物化學反應預測[11-12]、藥物化學合成[13-14]和藥物分子優化[15]等領域得到了廣泛應用。Transformer預訓練語言模型利用巨大的無標記語料庫來學習單詞和句子的表征,然后使用相對較小的數據集對預訓練模型進行微調以適應下游任務[16-17]。與傳統藥物化學描述符輸入深度學習預測模型不同的是,文獻[11-15]將藥物的簡化分子線性輸入規范(simplified molecular input line entry system,SMILES)[18]作為文本數據輸入模型。在藥物化學反應預測領域,Schwaller等[11]提出了一種基于Transformer結構的Molecular Transformer模型,通過貝葉斯深度學習訓練方法預測藥物化學反應并得到了較高的準確性。Wang等[12]在Transformer基礎上引入BERT半監督模型SMILES-BERT,通過微調使預訓練模型應用到不同的分子特性預測任務中。在藥物化學合成領域,Tetko等[13]提出了一種使用增強NLP Transformer模型的方法以確定化學反應的可行性,并通過評估合成步驟的預測結果來提高合成規劃的效率。Liu等[19]嘗試將Transformer模型應用到藥物組合協同作用預測領域,提出了一個基于注意力機制的Transformer深度學習模型TranSynergy,提高了協同藥物組合預測的性能和可解釋性。但是TranSynergy模型的輸入數據是O’Neil數據集中兩個藥物特征與一個細胞系特征組成的三元張量,由于輸入數據不是文本形式,所以模型主體部分的Transformer編解碼結構沒有使用詞向量嵌入和位置編碼,使得模型失去了理解藥物分子結構與藥物組合序列中分子距離特征的能力。
為了克服上述局限性,我們提出了一種以藥物的文本數據SMILES為輸入,以Transformer為預訓練模型的藥物協同作用預測方法——SMILESynergy。首先將藥物分子用SMILES表示,通過SMILES Enumeration對藥物組合進行數據增強,預訓練模型采用包括詞向量嵌入與位置編碼的Transformer模型以完成對藥物數據的編碼,在下游任務中連接一個多層感知器(multilayer perceptron,MLP)對藥物組合協同作用的回歸與分類任務進行預測。我們在O’Neil和NCI-ALMANAC數據集上對模型性能進行了驗證,并與DeepSynergy[5]、MulinputSynergy[8]等模型進行了比較。
1 數據集
本論文在兩個數據集上驗證SMILESynergy模型的性能。第一個數據集是默克公司制作的高通量藥物組合篩選數據集[6],文中稱為O’Neil數據集。該數據集包含583個藥物組合作用在39個癌癥細胞系上的22 737條實驗數據。Preuer等[5]在上述高通量篩選數據集上使用Combenefit[20]批量處理模式計算Loewe Additivity 值量化藥物組合的協同作用值,并將它作為回歸指標;在分類實驗中,我們采用文獻[5]中的閾值30進行分類,當藥物組合協同作用預測分數大于30時認為具有協同作用,反之無協同作用。
第二個數據集是NCI-ALMANAC數據集[21],包含了5 232個藥物對在NCI60的每個細胞系的組合活性,它可以用來發現那些與單一藥物相比具有更強生長抑制作用的藥物組合。在NCI-ALMANAC數據集中,ComboScore表示藥物組合活性:正值表示藥物組合具有協同作用,負值表示藥物組合無協同作用;PercentGrowth表示單個藥物或藥物組合的抗癌活性。我們用PercentGrowth和ComboScore分別作為回歸指標和分類指標對模型進行訓練與測試。
2 模型
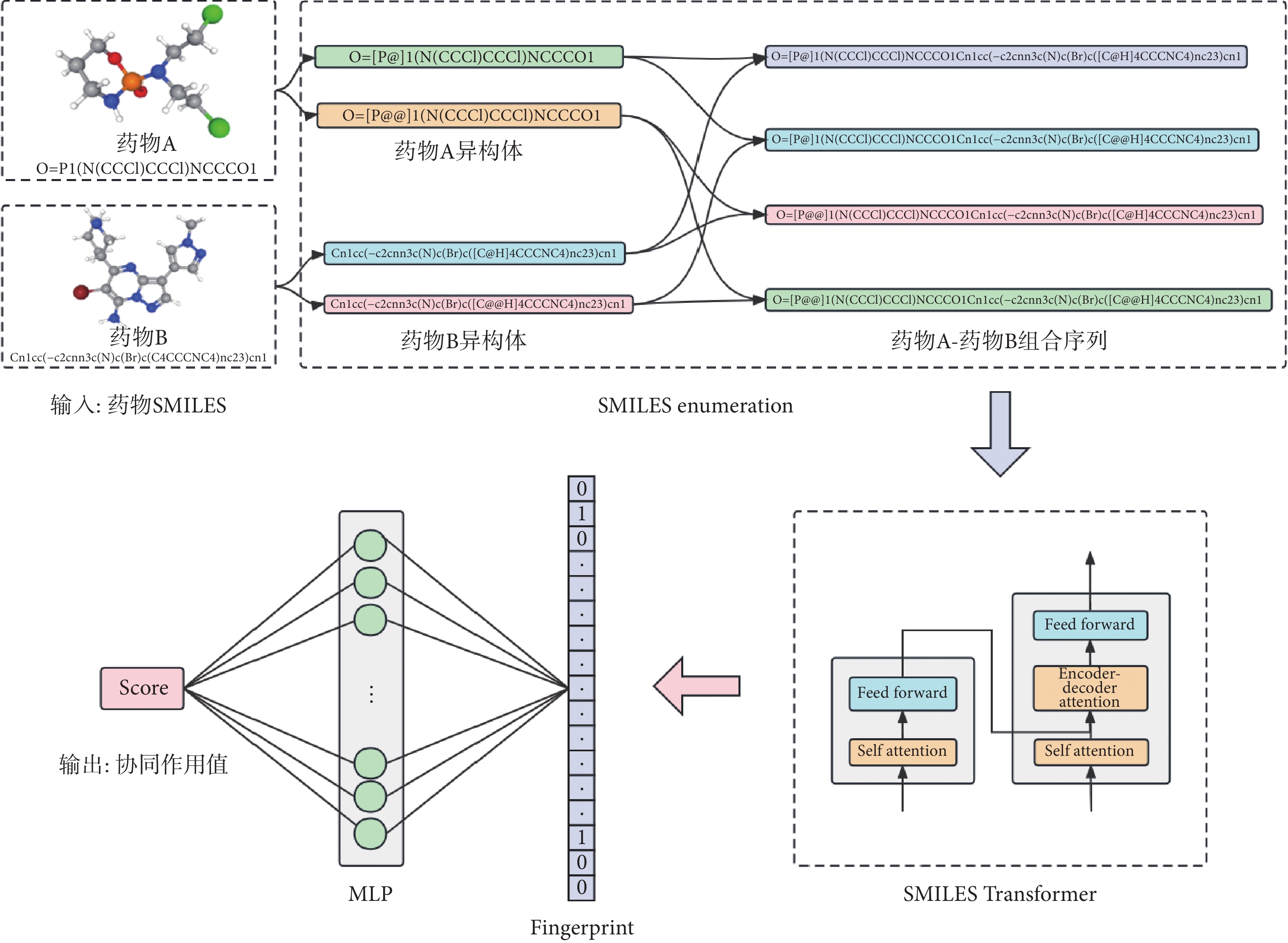
本模型主要由三部分構成,如圖1所示。第一部分是SMILES Enumeration生成藥物異構體,每種藥物不超過5個;第二部分是用于編解碼詞向量的Transformer模型,包含SMILES文件的詞嵌入、位置編碼和注意力機制;第三部分由MLP構成,對Transformer編碼的指紋文件(fingerprint)進行回歸預測,得到協同作用值表示模型的預測結果。
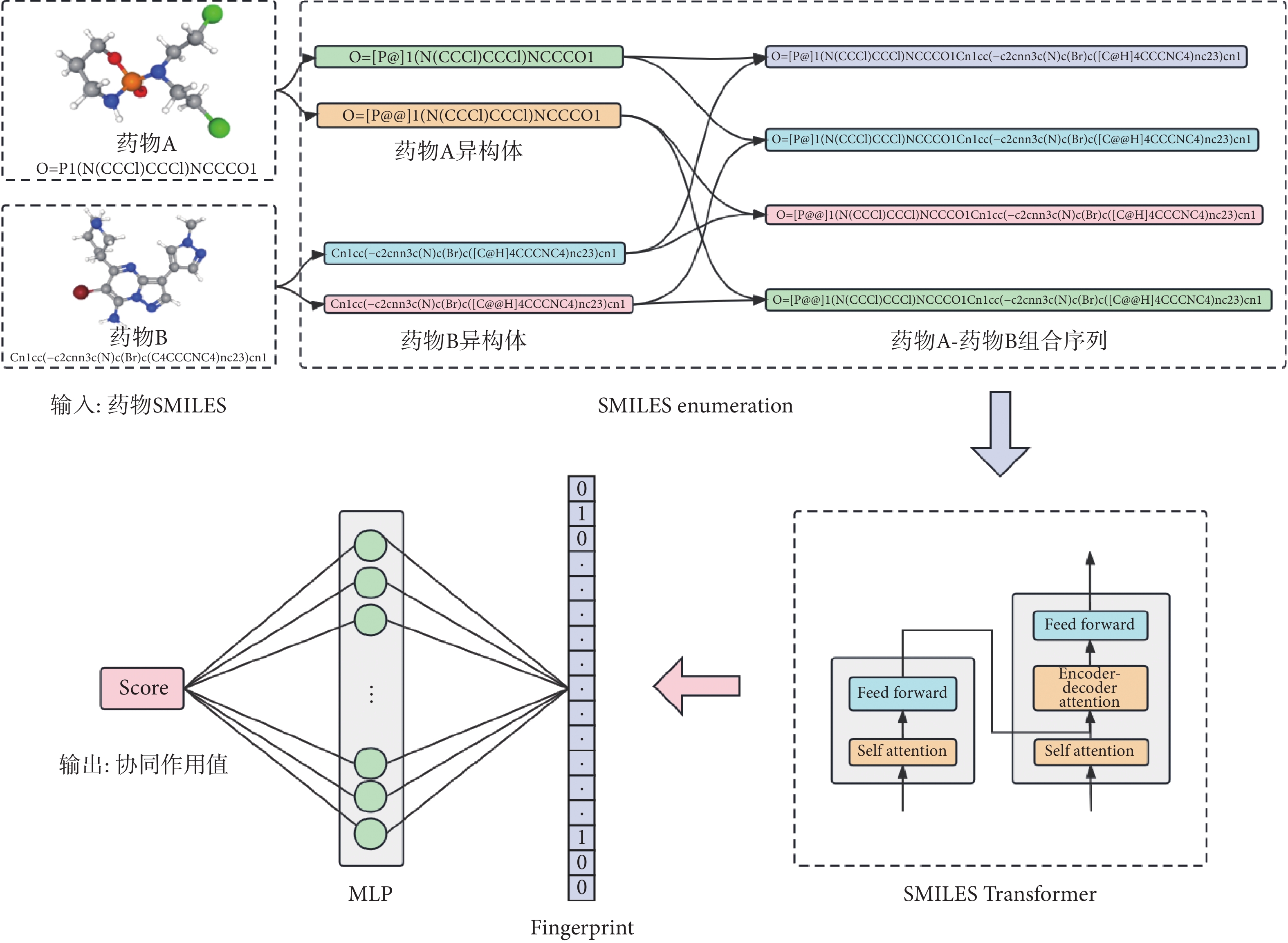 圖1
SMILESynergy 模型結構圖
Figure1.
Structure of SMILESynergy model
圖1
SMILESynergy 模型結構圖
Figure1.
Structure of SMILESynergy model
2.1 藥物的SMILES表示及SMILES Enumeration數據增強
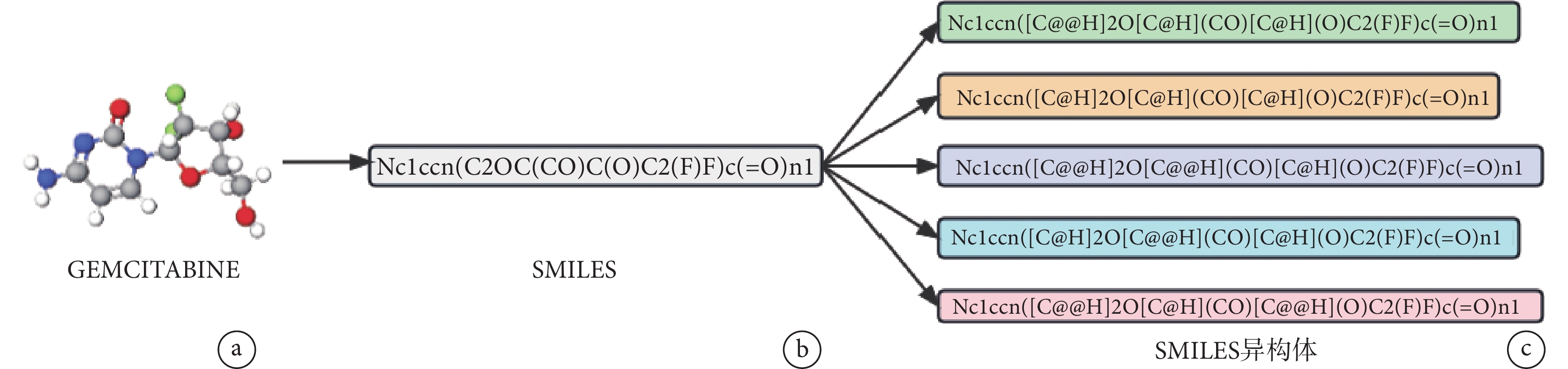
由于藥物的物理化學性質與藥物的分子排列組合方式和空間結構有關,并且Transformer模型在自然語言處理領域中通常以文本序列作為輸入數據[10],因此我們采用SMILES表征藥物。例如結構為C9H11F2N3O4的吉西他濱(GEMCITABINE)的SMILES表示(見圖2a)和三維分子結構(見圖2b),其中字母如C、N一般代表原子,符號如―、=、#代表化學鍵。考慮到O’Neil數據集中藥物對的數量僅有583個,我們對數據進行增強處理以提高模型的預測性能。SMILES可以表示藥物分子不同的異構體,包括構象異構體和位置異構體。我們利用RDKit[22]中EnumerateStereoisomers方法枚舉不同的SMILES獲得藥物分子的異構體來達到數據增強的目的。例如吉西他濱的分子異構體如圖2c所示。
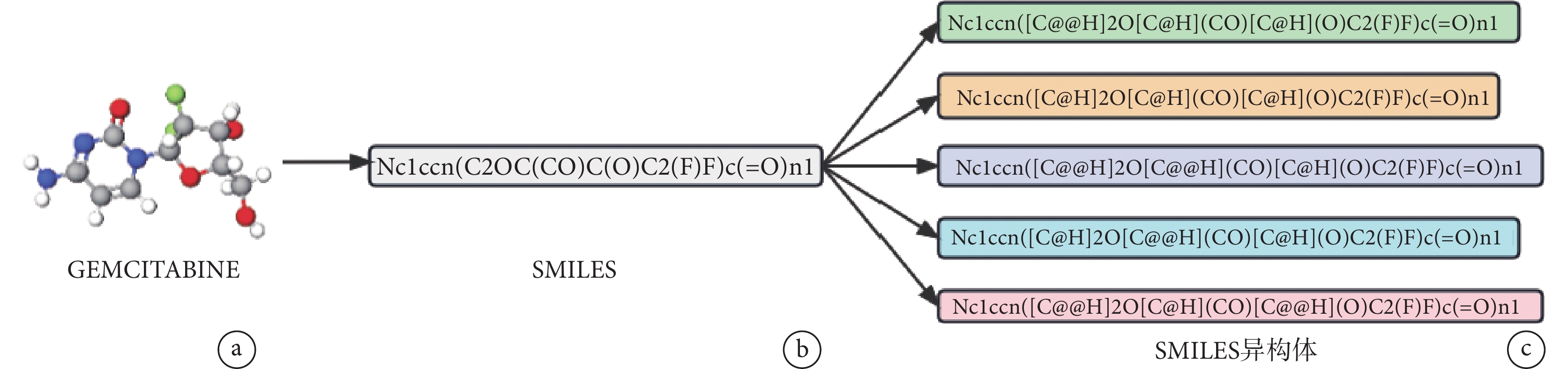 圖2
藥物吉西他濱
圖2
藥物吉西他濱
a. 三維分子結構;b. SMILES 表示;c. SMILES異構體
Figure2. Gemcitabinea. three-dimensional molecular structure of gemcitabine; b. SMILES representation; c. SMILES isomers
2.2 位置編碼與注意力機制
當SMILES Enumeration增強后的數據傳入Transformer模型時,我們對數據進行詞嵌入(Embedding)操作獲得藥物的詞嵌入向量。詞嵌入向量表示SMILES序列中的每個藥物分子映射到高維向量空間中的向量。為了進一步考慮藥物序列中每個分子的位置信息,我們將藥物分子的位置信息編碼之后添加到詞嵌入向量。位置編碼表示藥物分子在藥物序列中的位置或者功能團在藥物序列中的位置等,這樣模型就可以更好地利用藥物的物理化學性質和結構信息預測藥物的協同作用。Transformer中的位置編碼是由一組三角函數計算而來的,遵循如下公式:
 |
 |
其中 代表位置編碼(position encoding),
代表位置編碼(position encoding), 表示藥物分子在藥物序列中的位置,當
表示藥物分子在藥物序列中的位置,當  為偶數時使用正弦函數進行編碼,當
為偶數時使用正弦函數進行編碼,當  為奇數時使用余弦函數進行編碼。
為奇數時使用余弦函數進行編碼。 代表詞嵌入向量的維度,
代表詞嵌入向量的維度, 代表位于0到第
代表位于0到第  每一維元素都需要對輸入序列進行編碼。于是根據上述公式,我們可以得到藥物序列中第
每一維元素都需要對輸入序列進行編碼。于是根據上述公式,我們可以得到藥物序列中第  位置藥物分子的
位置藥物分子的  維位置向量。
維位置向量。
注意力機制是對藥物分子不同特征之間的相互作用進行編碼的模塊,它將一個Query和一組Key、Value鍵值對映射到一個輸出,其中Query表示待預測的藥物分子的查詢向量,用于捕獲其在特定任務中的特征表示;Key表示其他藥物分子的鍵向量,用于計算待預測藥物與其他藥物之間的相似性;Value表示其他藥物分子的值向量,用于加權計算其他藥物與待預測藥物的相似性得分。注意力的計算公式如下:
 |
 |
其中 是藥物特征矩陣,
是藥物特征矩陣, ,
, ,
, 分別對應Query、Key和Value的權重矩陣。
分別對應Query、Key和Value的權重矩陣。 是一個縮放因子,Z是注意力層的輸出。
是一個縮放因子,Z是注意力層的輸出。
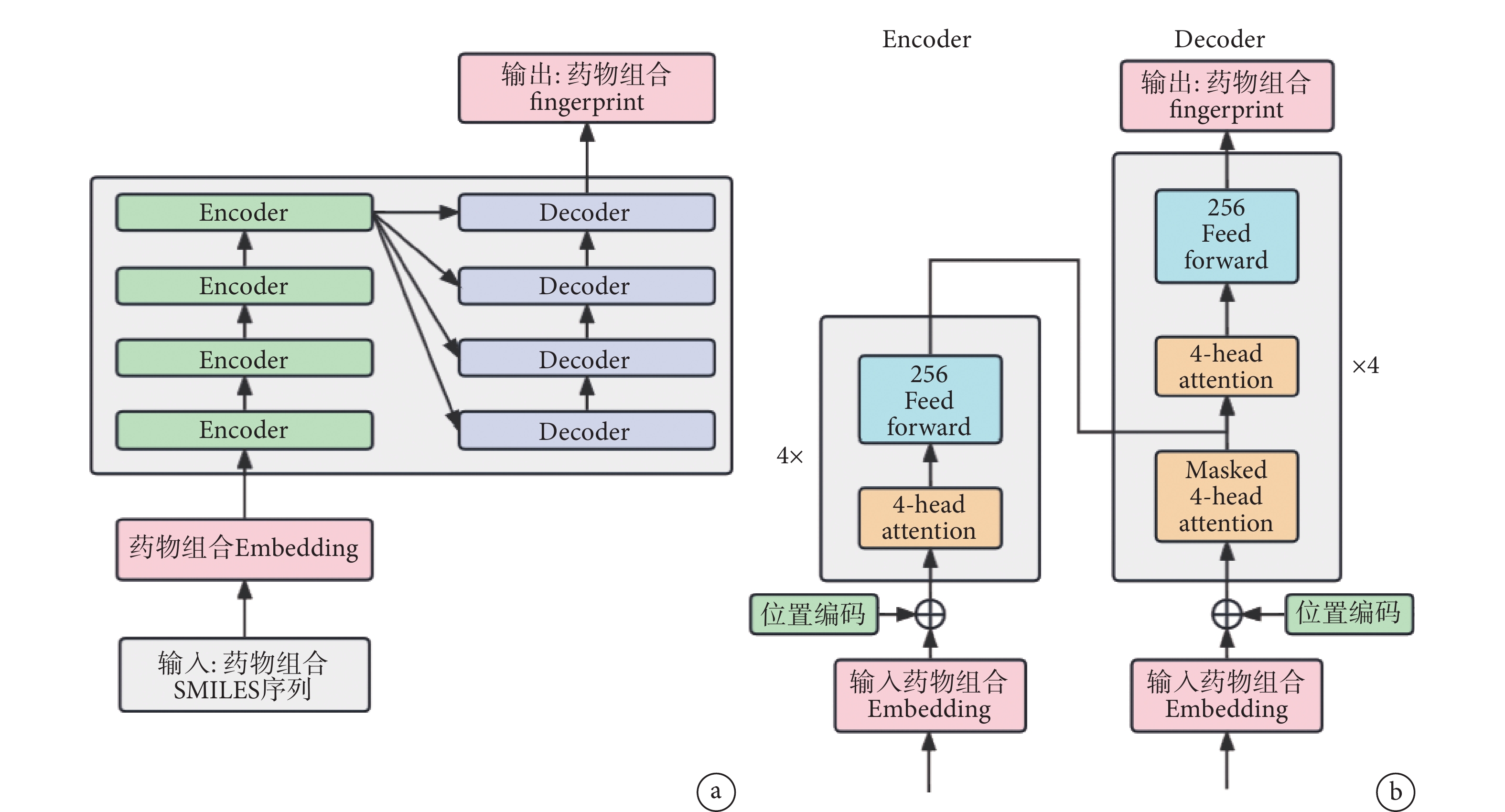
2.3 SMILES Transformer模型架構
我們的預訓練模型采用4層Encoder-Decoder的Transformer模型,模型整體架構如圖3a所示,輸入的藥物序列首先經過Embedding變為詞向量,并將位置編碼添加到詞嵌入向量中,隨后進入由4個編碼器與4個解碼器構成的Transformer結構。Transformer編碼器和解碼器內部細節如圖3b所示,Embedding后的藥物數據經過位置編碼進入到編碼器,編碼器由多個相同的層組成,每層包括一個多頭注意力層和一個全連接網絡層。解碼器也由多個相同的層組成,每層包括三個子層:多頭自注意力子層、編碼器-解碼器注意力子層和全連接前饋神經網絡子層。最終模型輸出1 024維的藥物fingerprint文件。
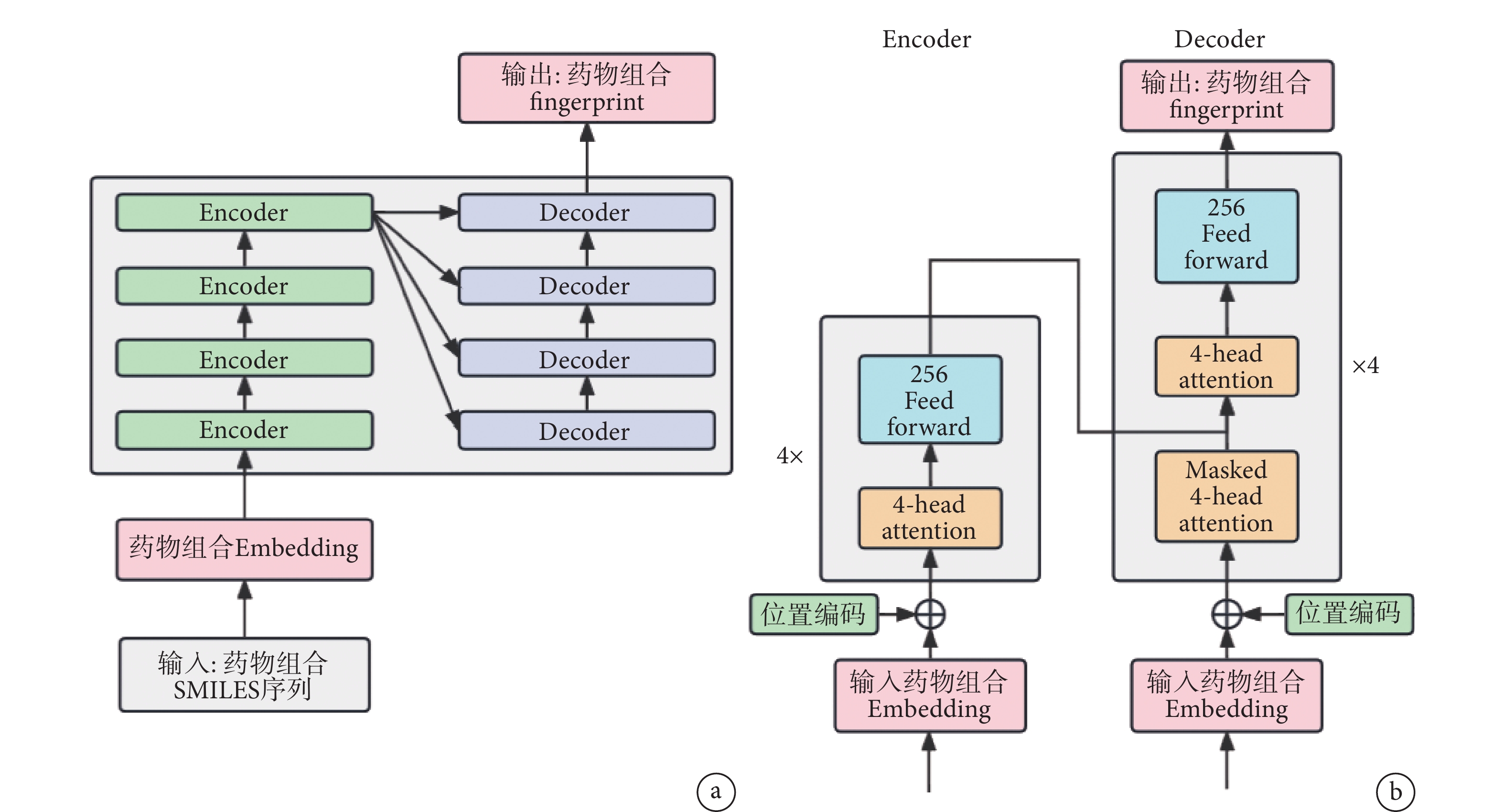 圖3
SMILES Transformer模型
圖3
SMILES Transformer模型
a. 模型整體架構;b. 模型內部細節
Figure3. SMILES Transformer modela. overall architecture of the model; b. internal details of the model
我們對ChEMBL24數據集(藥物化學和藥理學的數據集[23])中861 000個無標簽的SMILES隨機抽取進行預訓練,其架構參數及訓練超參數來自文獻[12]。在訓練中,使用Adam優化器最小化輸入SMILES和輸出概率之間的交叉熵。訓練過程中損失值逐漸下降,同時困惑度也在不斷減小,訓練了5個epoch后,模型達到收斂狀態,困惑度達到了1.0左右,表明模型能夠很好地預測出藥物組合序列中的每個藥物分子。相比原始Transformer[10],SMILES Transformer模型的參數由6 500萬下降至400萬左右,模型的收斂速度得到了提升。在下游任務中,我們利用SMILES Transformer編碼生成藥物的1 024維fingerprint數據輸入MLP,MLP的迭代次數max_iter設置為1 000,其他默認超參數與Scikit-learn[24]相同。隨后將數據集按4∶1隨機拆分為訓練集和測試集,進行最終的回歸訓練。SMILESynergy模型的偽代碼詳見附件1。
3 結果與討論
3.1 SMILESynergy在O’Neil數據集的性能評估
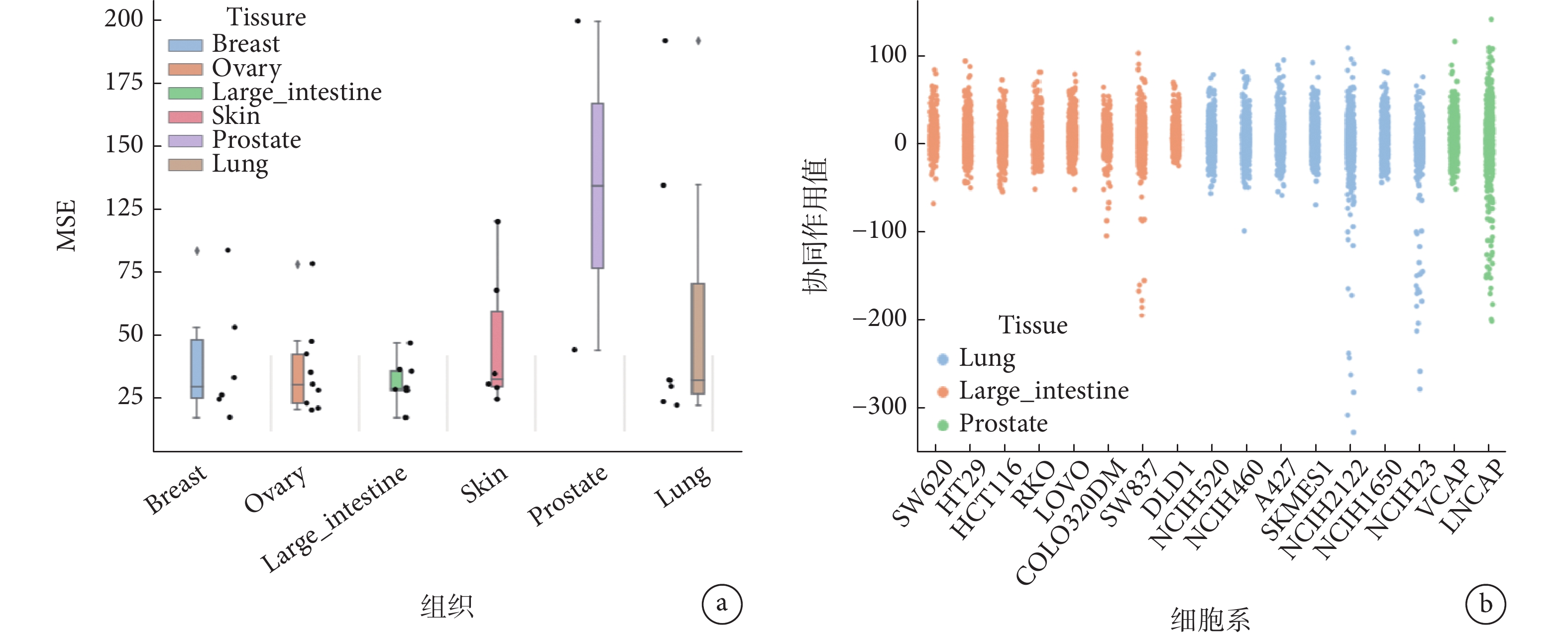
我們從回歸性能和分類性能兩方面對模型進行評估。回歸性能分析的主要指標是平均平方誤差(mean square error,MSE)、平均絕對誤差(mean absolute error ,MAE)、可釋方差值(explained variation score,EVS)、皮爾遜相關系數(Pearson)和R2(r-squared)。表1中給出了SMILESynergy模型在39個細胞系中的預測結果。
由表1可以看出,MSE的平均值降低到了100以內,在測試的39個細胞系中,各評價指標的中位數都非常接近平均值,說明我們的模型在大多數的細胞系中都具有良好的穩定性。但MSE的最小值與最大值相差約180,為了探究這一原因,根據器官組織分布,我們將細胞系的MSE包點圖和箱型圖進行了可視化,如圖4a所示,可以看出在乳腺(BREAST)、卵巢(OVARY)與肺(LUNG)三個組織中出現了三個異常值;導致MSE相差較大的異常情況主要出現在前列腺組織(PROSTATE)中,而前列腺組織涉及的細胞系只有兩個,因此數據平均值產生了較大的偏差。
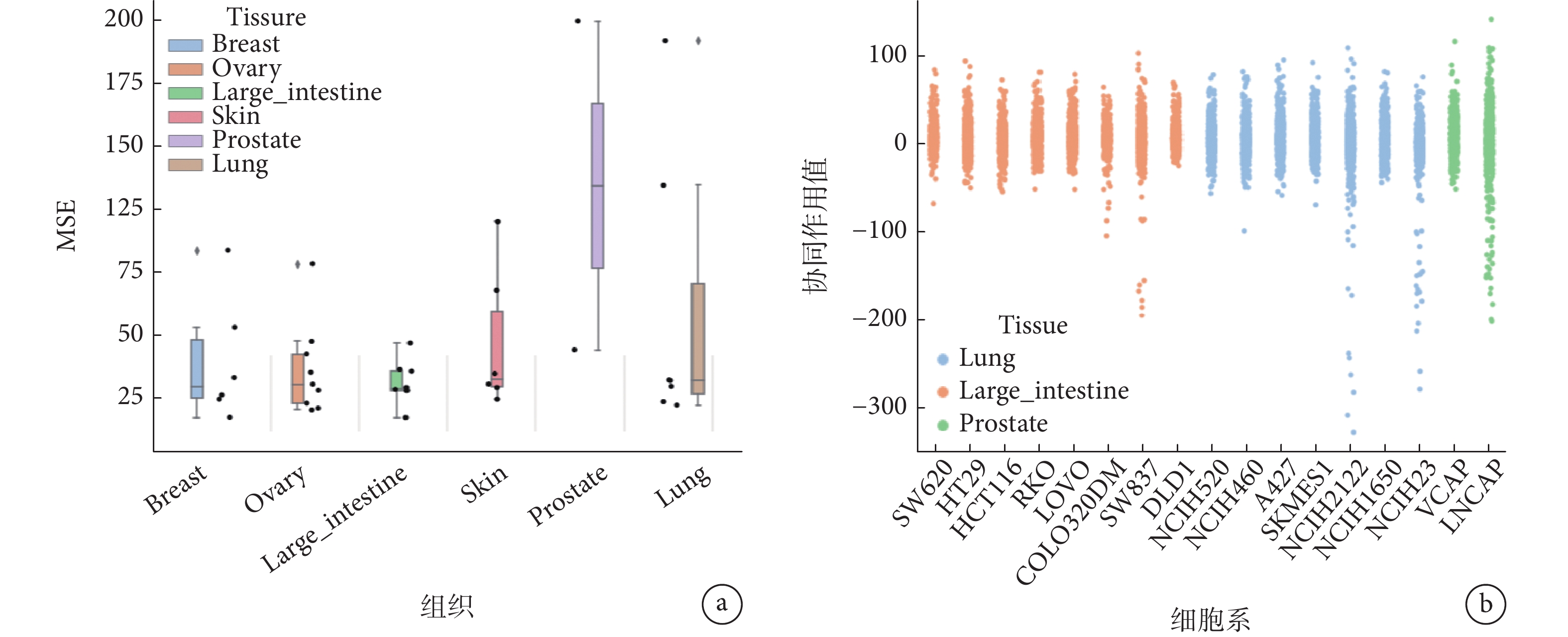 圖4
SMILESynergy在O’Neil數據集上回歸性能分析
圖4
SMILESynergy在O’Neil數據集上回歸性能分析
a. 各器官組織對應細胞系的MSE包點圖和箱型圖;b. 大腸、前列腺與肺組織的所有細胞系的協同值散點圖
Figure4. SMILESynergy regression performance analysis on the O’Neil dataseta. MSE beeswarm plots and box plots of corresponding cell lines for each organ tissue; b. scatter plots of synergy values of all cell lines for large intestine, prostate and lung tissues
為了進一步分析不同細胞系下預測結果的差異,我們選取圖4a中預測效果最好的大腸組織(LARGE_INTESTINE),以及預測效果較差的前列腺組織(PROSTATE)和肺組織(LUNG),對三個組織中所有細胞系對應的藥物組合協同作用值進行了可視化,得到散點圖4b。從圖4b可以看出,前列腺組織的LNCAP細胞系和肺組織的NCIH2122、NCIH23細胞系上的藥物組合協同作用值的分布較為分散,相較于其他細胞系有大量的數據分布在―100以下,藥物組合之間協同作用值的離散程度比較大,導致模型在訓練過程中無法有效學習藥物組合數據的特征規律,使得整體的MSE出現了較大范圍的波動。
在分類方面,我們采用準確率(accuracy,ACC)、精確率(precision,PREC)、敏感性(sensitivity,SENS)、特異性(specificity,SPEC)、F1分數(F1)、受試者工作特征曲線下的面積(area under curve of receiver operating characteristic,ROC_AUC)來衡量模型的分類性能。
從表2可以看到本文模型的平均準確率達到了0.97,特異性的平均值達到了0.98。同時注意到敏感性出現了一定幅度的波動,敏感性的最大值為0.93,小于特異性的最小值0.97,這說明SMILESynergy在學習預測藥物組合數據是否有協同作用的均衡性上有所欠缺。于是我們對O’Neil數據集中各組織藥物組合的類別數量分布進行了可視化(數據圖見附件2),發現O’Neil數據集中藥物組合具有協同作用和無協同作用的數據分布不均衡,無協同作用的藥物組合數量遠遠大于具有協同作用的藥物組合數量,導致敏感性與特異性出現了差異。
3.2 SMILESynergy 在NCI-ALMANAC數據集的性能評估
我們在NCI-ALMANAC數據集上對本文模型SMILESynergy進一步進行驗證,獲得了令人滿意的結果(詳見表3、表4)。在回歸方面,MAE和MSE的中位數均小于其平均值,R2、EVS和Pearson的中位數均高于平均值。在分類方面,ACC的中位數與平均值接近,而其他指標的中位數均高于平均值,這表明SMILESynergy在NCI-ALMANAC數據集的大多數細胞系上表現出了良好的穩定性。
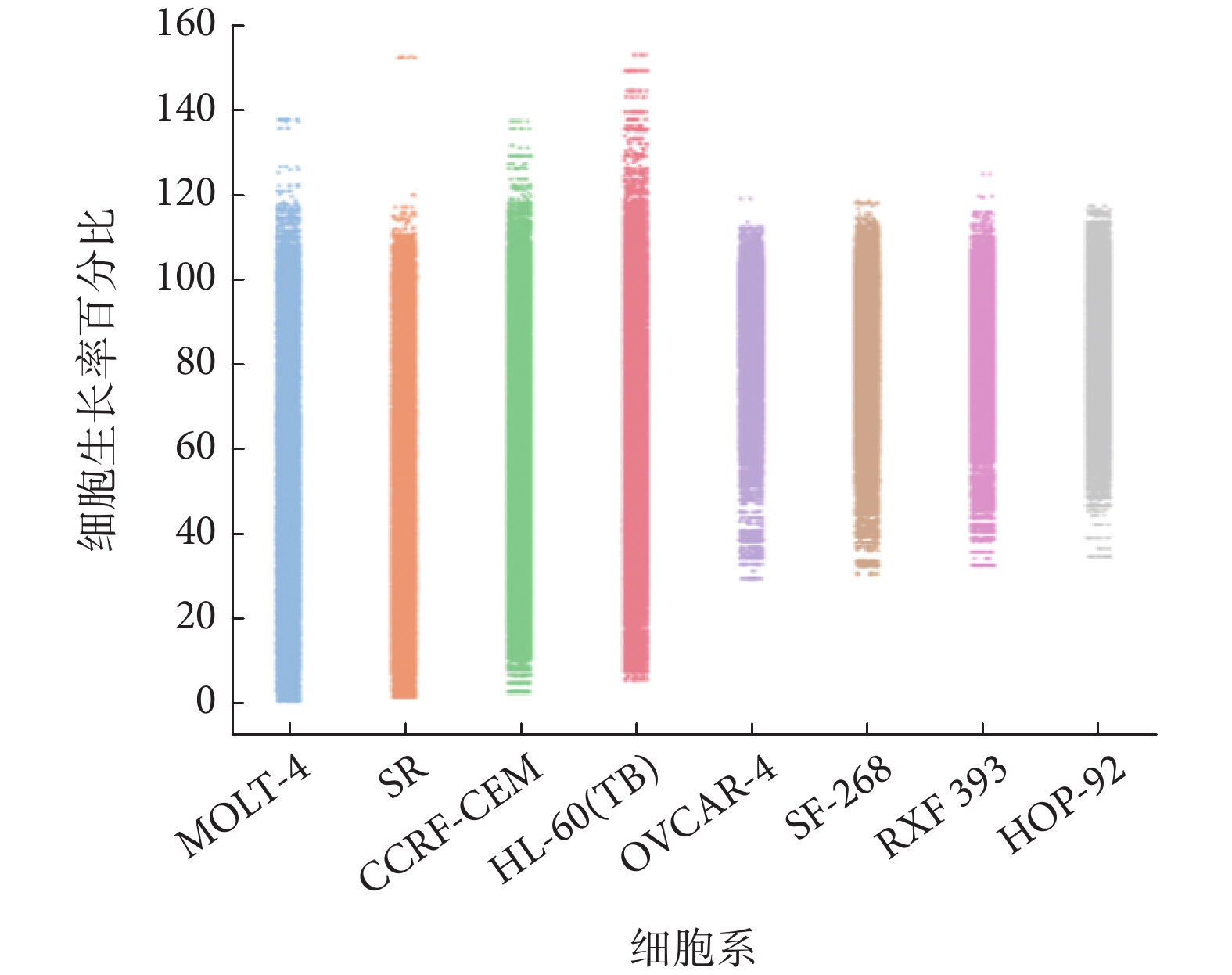
為了探究SMILESynergy在NCI-ALMANAC數據集中的預測性能,我們繪制了全局絕對誤差熱力圖(見附件3)。我們發現不同細胞系下的藥物組合數量存在差異,且大部分細胞系下的藥物組合的絕對誤差集中分布在10以內,這與實驗結果中MAE的平均值和中位數皆在10以內相一致。
在HL-60(TB)、SR、MOLT-4和CCRF-CEM這四個細胞系中,絕對誤差的波動相對較為明顯。我們對這四個細胞系及熱圖中預測效果較好的HOP-92、SF-268、RXF 393和OVCAR-4四個細胞系數據分布進行可視化分析,如圖5所示。結果顯示HL-60(TB)、SR、MOLT-4和CCRF-CEM四個細胞系的細胞生長率分布較為分散,使得模型難以學習藥物組合數據的特征規律,從而導致整體絕對誤差波動較大。
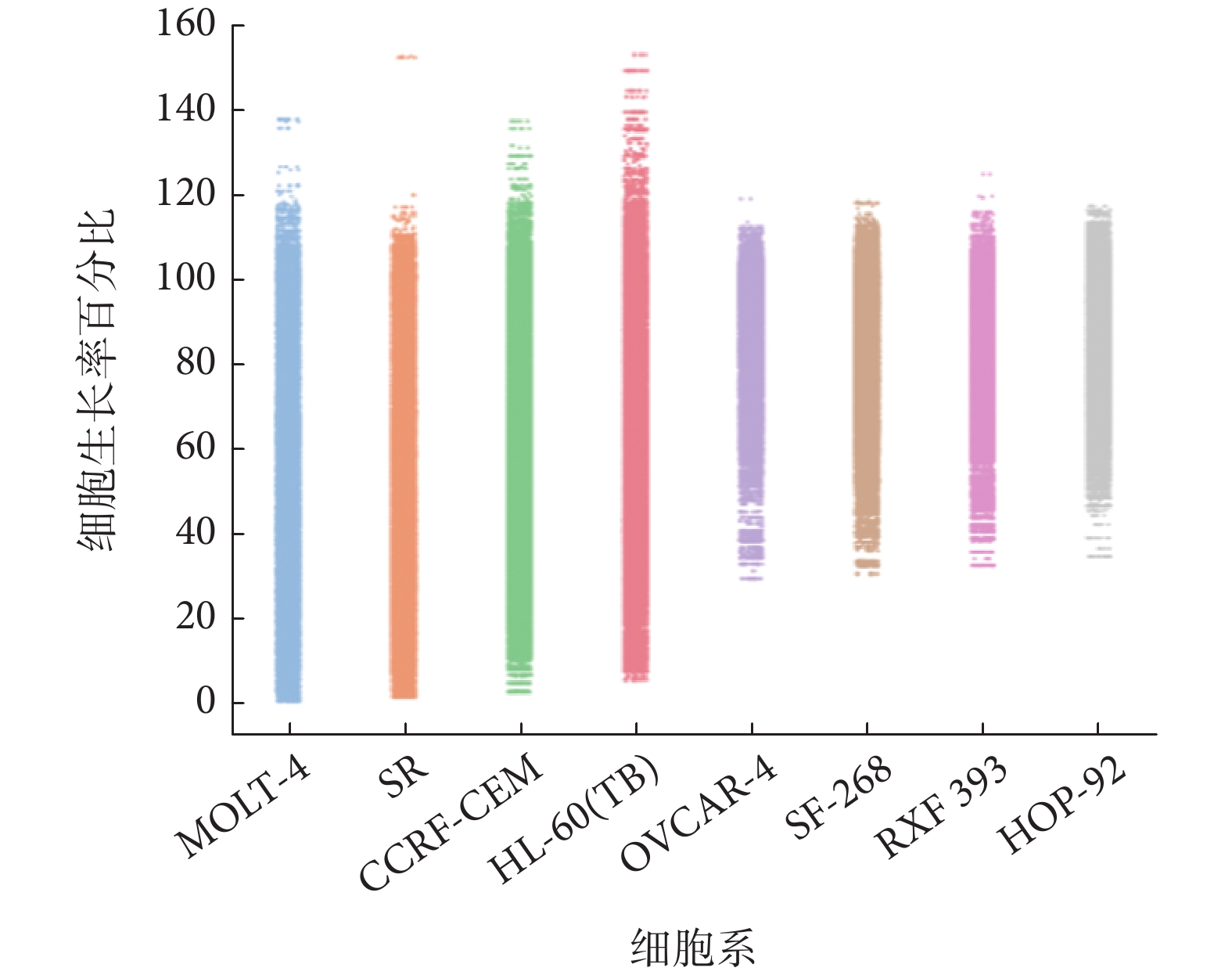 圖5
NCI-ALMANAC 數據集中部分細胞系的細胞生長率百分比散點圖
Figure5.
Scatter plot of cell growth rate percentage for selected cell lines in the NCI-ALMANAC dataset
圖5
NCI-ALMANAC 數據集中部分細胞系的細胞生長率百分比散點圖
Figure5.
Scatter plot of cell growth rate percentage for selected cell lines in the NCI-ALMANAC dataset
3.3 方法對比
為了驗證SMILESynergy模型的預測性能,我們在O’Neil數據集上與DeepSynergy模型[5]和MulinputSynergy模型[8]進行實驗對比。我們的模型將藥物轉換成SMILES文件作為輸入數據。DeepSynergy計算了藥物三種不同類型的化學特征:首先使用jCompoundMapper[25]生成半徑為6(ECFP_6)的擴展連接指紋計數,然后利用ChemoPy[26]計算藥物的物理化學性質,最后從文獻[27]中收集到一組亞結構毒團特征。MulinputSynergy則使用軟件alvaDesc計算藥物化合物的分子描述符,包括功能組、片段計數和藥效團等。
三個模型詳細的比較結果列于表5和表6。從表5中可以看出,SMILESynergy模型在預測抗癌藥協同作用的回歸任務中,相對于MulinputSynergy和DeepSynergy模型,具有更小的MSE和MAE,以及更高的EVS、Pearson和R2指標,這表明SMILESynergy模型能夠更準確地預測抗癌藥協同作用的強度,并且預測結果與實際值之間的相關性更強。從表6中可以看出,SMILESynergy模型在預測抗癌藥協同作用的分類任務中,相對于MulinputSynergy和DeepSynergy模型,具有更高的ACC、PREC、SENS、F1和ROC_AUC指標,這表明SMILESynergy模型不僅能夠準確地預測抗癌藥協同作用的類別,還能夠在預測結果的精度和召回率之間做到更好的平衡。
3.4 SMILES Enumeration消融實驗
我們將SMILESynergy模型進行消融實驗研究來分析SMILES Enumeration模塊對抗癌藥物協同作用預測的影響,分別評估了無SMILES Enumeration以及加入SMILES Enumeration后的影響,對比結果如表7所示,各指標皆為兩模型在各自細胞系上的平均值。由表7可以看出,在加入SMILES Enumeration之后,模型在兩個數據集上的回歸與分類性能都得到了一定程度的提升。原因可能是經過SMILES Enumeration之后使得模型對藥物組合數據的特征進行了更多的訓練與學習,從而提升了自身的魯棒性。
4 結論
我們提出了一種新的抗癌藥物協同作用預測模型SMILESynergy。模型以藥物的SMILES作為輸入,并利用異構體數據進行數據增強,然后采用預訓練的Transformer模型編碼藥物組合數據,最后通過連接MLP完成對藥物組合協同作用的預測。在O’Neil數據集上,SMILESynergy優于其他模型,具有更好的預測性能,可幫助研究人員快速篩選最優藥物組合,提高癌癥治療效果。數據及工程文件可在https://github.com/unclestrong/SMILESynergy獲取。
重要聲明
利益沖突聲明:本文全體作者均聲明不存在利益沖突。
作者貢獻聲明:張立強主要負責實驗方案設計、代碼編寫與調試、實驗結果分析與論文撰寫,秦玉芳主要負責算法咨詢與建議、實驗結果分析、論文撰寫與指導,陳明作者為論文提供了資助和支持。
本文附件見本刊網站的電子版本(biomedeng.cn)。
0 引言
癌癥是一種高度異質性的疾病,不同類型的癌癥具有不同的遺傳變異和表觀遺傳變異[1],導致不同個體對藥物的反應和耐藥性[2]也不同,因此選擇最優的藥物組合來提高治療效果是癌癥治療的一個重要挑戰。傳統的化療藥物研究方法需要通過大量的臨床試驗進行藥物篩選和劑量確定,耗費時間和資源,并且可能使患者面臨不必要甚至有害的治療[3]。而且由于癌癥的異質性和個體差異性,不同患者對同一種藥物的反應和副作用也不同,使得制定治療方案更加困難。因此利用計算模型來探索高通量篩選[4]實驗產生的藥物組合數據具有重要意義。
近年來研究人員運用深度學習在抗癌藥物協同作用預測的研究中做了大量的工作。如Preuer等[5]提出的DeepSynergy模型,以O’Neil數據集[6]上的藥物化合物以及細胞系基因表達信息作為輸入,連接一個全連接前饋神經網絡(fully forward neural network,FFNN),得到了不錯的抗癌藥物組合協同作用預測結果。在藥物化合物以及細胞系基因表達信息的基礎上,Zhang等[7]加入細胞系的拷貝數和基因突變數據作為輸入,并提出了基于自編碼器(auto encoder)的深度學習模型AuDNNsynergy來預測具有協同作用的藥物組合。在文獻[7]輸入數據的基礎上,陳希等[8]通過一維卷積層對輸入數據進行降維預處理,進而提出了MulinputSynergy藥物預測模型,并在模型中加入殘差前饋神經層,最終提升了藥物組合預測效果。Sun等[9]在O’Neil數據集的藥物數據上引入張量分解方法進行預處理,然后將分解的結果作為特征用于訓練深度神經網絡(deep neural network,DNN)模型以預測藥物對的協同效應。
鑒于近年來深度學習在自然語言處理(natural language process,NLP)領域的發展,以Transformer[10]為代表的預訓練語言模型加下游任務的方案在藥物化學反應預測[11-12]、藥物化學合成[13-14]和藥物分子優化[15]等領域得到了廣泛應用。Transformer預訓練語言模型利用巨大的無標記語料庫來學習單詞和句子的表征,然后使用相對較小的數據集對預訓練模型進行微調以適應下游任務[16-17]。與傳統藥物化學描述符輸入深度學習預測模型不同的是,文獻[11-15]將藥物的簡化分子線性輸入規范(simplified molecular input line entry system,SMILES)[18]作為文本數據輸入模型。在藥物化學反應預測領域,Schwaller等[11]提出了一種基于Transformer結構的Molecular Transformer模型,通過貝葉斯深度學習訓練方法預測藥物化學反應并得到了較高的準確性。Wang等[12]在Transformer基礎上引入BERT半監督模型SMILES-BERT,通過微調使預訓練模型應用到不同的分子特性預測任務中。在藥物化學合成領域,Tetko等[13]提出了一種使用增強NLP Transformer模型的方法以確定化學反應的可行性,并通過評估合成步驟的預測結果來提高合成規劃的效率。Liu等[19]嘗試將Transformer模型應用到藥物組合協同作用預測領域,提出了一個基于注意力機制的Transformer深度學習模型TranSynergy,提高了協同藥物組合預測的性能和可解釋性。但是TranSynergy模型的輸入數據是O’Neil數據集中兩個藥物特征與一個細胞系特征組成的三元張量,由于輸入數據不是文本形式,所以模型主體部分的Transformer編解碼結構沒有使用詞向量嵌入和位置編碼,使得模型失去了理解藥物分子結構與藥物組合序列中分子距離特征的能力。
為了克服上述局限性,我們提出了一種以藥物的文本數據SMILES為輸入,以Transformer為預訓練模型的藥物協同作用預測方法——SMILESynergy。首先將藥物分子用SMILES表示,通過SMILES Enumeration對藥物組合進行數據增強,預訓練模型采用包括詞向量嵌入與位置編碼的Transformer模型以完成對藥物數據的編碼,在下游任務中連接一個多層感知器(multilayer perceptron,MLP)對藥物組合協同作用的回歸與分類任務進行預測。我們在O’Neil和NCI-ALMANAC數據集上對模型性能進行了驗證,并與DeepSynergy[5]、MulinputSynergy[8]等模型進行了比較。
1 數據集
本論文在兩個數據集上驗證SMILESynergy模型的性能。第一個數據集是默克公司制作的高通量藥物組合篩選數據集[6],文中稱為O’Neil數據集。該數據集包含583個藥物組合作用在39個癌癥細胞系上的22 737條實驗數據。Preuer等[5]在上述高通量篩選數據集上使用Combenefit[20]批量處理模式計算Loewe Additivity 值量化藥物組合的協同作用值,并將它作為回歸指標;在分類實驗中,我們采用文獻[5]中的閾值30進行分類,當藥物組合協同作用預測分數大于30時認為具有協同作用,反之無協同作用。
第二個數據集是NCI-ALMANAC數據集[21],包含了5 232個藥物對在NCI60的每個細胞系的組合活性,它可以用來發現那些與單一藥物相比具有更強生長抑制作用的藥物組合。在NCI-ALMANAC數據集中,ComboScore表示藥物組合活性:正值表示藥物組合具有協同作用,負值表示藥物組合無協同作用;PercentGrowth表示單個藥物或藥物組合的抗癌活性。我們用PercentGrowth和ComboScore分別作為回歸指標和分類指標對模型進行訓練與測試。
2 模型
本模型主要由三部分構成,如圖1所示。第一部分是SMILES Enumeration生成藥物異構體,每種藥物不超過5個;第二部分是用于編解碼詞向量的Transformer模型,包含SMILES文件的詞嵌入、位置編碼和注意力機制;第三部分由MLP構成,對Transformer編碼的指紋文件(fingerprint)進行回歸預測,得到協同作用值表示模型的預測結果。
 圖1
SMILESynergy 模型結構圖
Figure1.
Structure of SMILESynergy model
圖1
SMILESynergy 模型結構圖
Figure1.
Structure of SMILESynergy model
2.1 藥物的SMILES表示及SMILES Enumeration數據增強
由于藥物的物理化學性質與藥物的分子排列組合方式和空間結構有關,并且Transformer模型在自然語言處理領域中通常以文本序列作為輸入數據[10],因此我們采用SMILES表征藥物。例如結構為C9H11F2N3O4的吉西他濱(GEMCITABINE)的SMILES表示(見圖2a)和三維分子結構(見圖2b),其中字母如C、N一般代表原子,符號如―、=、#代表化學鍵。考慮到O’Neil數據集中藥物對的數量僅有583個,我們對數據進行增強處理以提高模型的預測性能。SMILES可以表示藥物分子不同的異構體,包括構象異構體和位置異構體。我們利用RDKit[22]中EnumerateStereoisomers方法枚舉不同的SMILES獲得藥物分子的異構體來達到數據增強的目的。例如吉西他濱的分子異構體如圖2c所示。
 圖2
藥物吉西他濱
圖2
藥物吉西他濱
a. 三維分子結構;b. SMILES 表示;c. SMILES異構體
Figure2. Gemcitabinea. three-dimensional molecular structure of gemcitabine; b. SMILES representation; c. SMILES isomers
2.2 位置編碼與注意力機制
當SMILES Enumeration增強后的數據傳入Transformer模型時,我們對數據進行詞嵌入(Embedding)操作獲得藥物的詞嵌入向量。詞嵌入向量表示SMILES序列中的每個藥物分子映射到高維向量空間中的向量。為了進一步考慮藥物序列中每個分子的位置信息,我們將藥物分子的位置信息編碼之后添加到詞嵌入向量。位置編碼表示藥物分子在藥物序列中的位置或者功能團在藥物序列中的位置等,這樣模型就可以更好地利用藥物的物理化學性質和結構信息預測藥物的協同作用。Transformer中的位置編碼是由一組三角函數計算而來的,遵循如下公式:
 |
 |
其中 代表位置編碼(position encoding),
代表位置編碼(position encoding), 表示藥物分子在藥物序列中的位置,當
表示藥物分子在藥物序列中的位置,當  為偶數時使用正弦函數進行編碼,當
為偶數時使用正弦函數進行編碼,當  為奇數時使用余弦函數進行編碼。
為奇數時使用余弦函數進行編碼。 代表詞嵌入向量的維度,
代表詞嵌入向量的維度, 代表位于0到第
代表位于0到第  每一維元素都需要對輸入序列進行編碼。于是根據上述公式,我們可以得到藥物序列中第
每一維元素都需要對輸入序列進行編碼。于是根據上述公式,我們可以得到藥物序列中第  位置藥物分子的
位置藥物分子的  維位置向量。
維位置向量。
注意力機制是對藥物分子不同特征之間的相互作用進行編碼的模塊,它將一個Query和一組Key、Value鍵值對映射到一個輸出,其中Query表示待預測的藥物分子的查詢向量,用于捕獲其在特定任務中的特征表示;Key表示其他藥物分子的鍵向量,用于計算待預測藥物與其他藥物之間的相似性;Value表示其他藥物分子的值向量,用于加權計算其他藥物與待預測藥物的相似性得分。注意力的計算公式如下:
 |
 |
其中 是藥物特征矩陣,
是藥物特征矩陣, ,
, ,
, 分別對應Query、Key和Value的權重矩陣。
分別對應Query、Key和Value的權重矩陣。 是一個縮放因子,Z是注意力層的輸出。
是一個縮放因子,Z是注意力層的輸出。
2.3 SMILES Transformer模型架構
我們的預訓練模型采用4層Encoder-Decoder的Transformer模型,模型整體架構如圖3a所示,輸入的藥物序列首先經過Embedding變為詞向量,并將位置編碼添加到詞嵌入向量中,隨后進入由4個編碼器與4個解碼器構成的Transformer結構。Transformer編碼器和解碼器內部細節如圖3b所示,Embedding后的藥物數據經過位置編碼進入到編碼器,編碼器由多個相同的層組成,每層包括一個多頭注意力層和一個全連接網絡層。解碼器也由多個相同的層組成,每層包括三個子層:多頭自注意力子層、編碼器-解碼器注意力子層和全連接前饋神經網絡子層。最終模型輸出1 024維的藥物fingerprint文件。
 圖3
SMILES Transformer模型
圖3
SMILES Transformer模型
a. 模型整體架構;b. 模型內部細節
Figure3. SMILES Transformer modela. overall architecture of the model; b. internal details of the model
我們對ChEMBL24數據集(藥物化學和藥理學的數據集[23])中861 000個無標簽的SMILES隨機抽取進行預訓練,其架構參數及訓練超參數來自文獻[12]。在訓練中,使用Adam優化器最小化輸入SMILES和輸出概率之間的交叉熵。訓練過程中損失值逐漸下降,同時困惑度也在不斷減小,訓練了5個epoch后,模型達到收斂狀態,困惑度達到了1.0左右,表明模型能夠很好地預測出藥物組合序列中的每個藥物分子。相比原始Transformer[10],SMILES Transformer模型的參數由6 500萬下降至400萬左右,模型的收斂速度得到了提升。在下游任務中,我們利用SMILES Transformer編碼生成藥物的1 024維fingerprint數據輸入MLP,MLP的迭代次數max_iter設置為1 000,其他默認超參數與Scikit-learn[24]相同。隨后將數據集按4∶1隨機拆分為訓練集和測試集,進行最終的回歸訓練。SMILESynergy模型的偽代碼詳見附件1。
3 結果與討論
3.1 SMILESynergy在O’Neil數據集的性能評估
我們從回歸性能和分類性能兩方面對模型進行評估。回歸性能分析的主要指標是平均平方誤差(mean square error,MSE)、平均絕對誤差(mean absolute error ,MAE)、可釋方差值(explained variation score,EVS)、皮爾遜相關系數(Pearson)和R2(r-squared)。表1中給出了SMILESynergy模型在39個細胞系中的預測結果。
由表1可以看出,MSE的平均值降低到了100以內,在測試的39個細胞系中,各評價指標的中位數都非常接近平均值,說明我們的模型在大多數的細胞系中都具有良好的穩定性。但MSE的最小值與最大值相差約180,為了探究這一原因,根據器官組織分布,我們將細胞系的MSE包點圖和箱型圖進行了可視化,如圖4a所示,可以看出在乳腺(BREAST)、卵巢(OVARY)與肺(LUNG)三個組織中出現了三個異常值;導致MSE相差較大的異常情況主要出現在前列腺組織(PROSTATE)中,而前列腺組織涉及的細胞系只有兩個,因此數據平均值產生了較大的偏差。
 圖4
SMILESynergy在O’Neil數據集上回歸性能分析
圖4
SMILESynergy在O’Neil數據集上回歸性能分析
a. 各器官組織對應細胞系的MSE包點圖和箱型圖;b. 大腸、前列腺與肺組織的所有細胞系的協同值散點圖
Figure4. SMILESynergy regression performance analysis on the O’Neil dataseta. MSE beeswarm plots and box plots of corresponding cell lines for each organ tissue; b. scatter plots of synergy values of all cell lines for large intestine, prostate and lung tissues
為了進一步分析不同細胞系下預測結果的差異,我們選取圖4a中預測效果最好的大腸組織(LARGE_INTESTINE),以及預測效果較差的前列腺組織(PROSTATE)和肺組織(LUNG),對三個組織中所有細胞系對應的藥物組合協同作用值進行了可視化,得到散點圖4b。從圖4b可以看出,前列腺組織的LNCAP細胞系和肺組織的NCIH2122、NCIH23細胞系上的藥物組合協同作用值的分布較為分散,相較于其他細胞系有大量的數據分布在―100以下,藥物組合之間協同作用值的離散程度比較大,導致模型在訓練過程中無法有效學習藥物組合數據的特征規律,使得整體的MSE出現了較大范圍的波動。
在分類方面,我們采用準確率(accuracy,ACC)、精確率(precision,PREC)、敏感性(sensitivity,SENS)、特異性(specificity,SPEC)、F1分數(F1)、受試者工作特征曲線下的面積(area under curve of receiver operating characteristic,ROC_AUC)來衡量模型的分類性能。
從表2可以看到本文模型的平均準確率達到了0.97,特異性的平均值達到了0.98。同時注意到敏感性出現了一定幅度的波動,敏感性的最大值為0.93,小于特異性的最小值0.97,這說明SMILESynergy在學習預測藥物組合數據是否有協同作用的均衡性上有所欠缺。于是我們對O’Neil數據集中各組織藥物組合的類別數量分布進行了可視化(數據圖見附件2),發現O’Neil數據集中藥物組合具有協同作用和無協同作用的數據分布不均衡,無協同作用的藥物組合數量遠遠大于具有協同作用的藥物組合數量,導致敏感性與特異性出現了差異。
3.2 SMILESynergy 在NCI-ALMANAC數據集的性能評估
我們在NCI-ALMANAC數據集上對本文模型SMILESynergy進一步進行驗證,獲得了令人滿意的結果(詳見表3、表4)。在回歸方面,MAE和MSE的中位數均小于其平均值,R2、EVS和Pearson的中位數均高于平均值。在分類方面,ACC的中位數與平均值接近,而其他指標的中位數均高于平均值,這表明SMILESynergy在NCI-ALMANAC數據集的大多數細胞系上表現出了良好的穩定性。
為了探究SMILESynergy在NCI-ALMANAC數據集中的預測性能,我們繪制了全局絕對誤差熱力圖(見附件3)。我們發現不同細胞系下的藥物組合數量存在差異,且大部分細胞系下的藥物組合的絕對誤差集中分布在10以內,這與實驗結果中MAE的平均值和中位數皆在10以內相一致。
在HL-60(TB)、SR、MOLT-4和CCRF-CEM這四個細胞系中,絕對誤差的波動相對較為明顯。我們對這四個細胞系及熱圖中預測效果較好的HOP-92、SF-268、RXF 393和OVCAR-4四個細胞系數據分布進行可視化分析,如圖5所示。結果顯示HL-60(TB)、SR、MOLT-4和CCRF-CEM四個細胞系的細胞生長率分布較為分散,使得模型難以學習藥物組合數據的特征規律,從而導致整體絕對誤差波動較大。
 圖5
NCI-ALMANAC 數據集中部分細胞系的細胞生長率百分比散點圖
Figure5.
Scatter plot of cell growth rate percentage for selected cell lines in the NCI-ALMANAC dataset
圖5
NCI-ALMANAC 數據集中部分細胞系的細胞生長率百分比散點圖
Figure5.
Scatter plot of cell growth rate percentage for selected cell lines in the NCI-ALMANAC dataset
3.3 方法對比
為了驗證SMILESynergy模型的預測性能,我們在O’Neil數據集上與DeepSynergy模型[5]和MulinputSynergy模型[8]進行實驗對比。我們的模型將藥物轉換成SMILES文件作為輸入數據。DeepSynergy計算了藥物三種不同類型的化學特征:首先使用jCompoundMapper[25]生成半徑為6(ECFP_6)的擴展連接指紋計數,然后利用ChemoPy[26]計算藥物的物理化學性質,最后從文獻[27]中收集到一組亞結構毒團特征。MulinputSynergy則使用軟件alvaDesc計算藥物化合物的分子描述符,包括功能組、片段計數和藥效團等。
三個模型詳細的比較結果列于表5和表6。從表5中可以看出,SMILESynergy模型在預測抗癌藥協同作用的回歸任務中,相對于MulinputSynergy和DeepSynergy模型,具有更小的MSE和MAE,以及更高的EVS、Pearson和R2指標,這表明SMILESynergy模型能夠更準確地預測抗癌藥協同作用的強度,并且預測結果與實際值之間的相關性更強。從表6中可以看出,SMILESynergy模型在預測抗癌藥協同作用的分類任務中,相對于MulinputSynergy和DeepSynergy模型,具有更高的ACC、PREC、SENS、F1和ROC_AUC指標,這表明SMILESynergy模型不僅能夠準確地預測抗癌藥協同作用的類別,還能夠在預測結果的精度和召回率之間做到更好的平衡。
3.4 SMILES Enumeration消融實驗
我們將SMILESynergy模型進行消融實驗研究來分析SMILES Enumeration模塊對抗癌藥物協同作用預測的影響,分別評估了無SMILES Enumeration以及加入SMILES Enumeration后的影響,對比結果如表7所示,各指標皆為兩模型在各自細胞系上的平均值。由表7可以看出,在加入SMILES Enumeration之后,模型在兩個數據集上的回歸與分類性能都得到了一定程度的提升。原因可能是經過SMILES Enumeration之后使得模型對藥物組合數據的特征進行了更多的訓練與學習,從而提升了自身的魯棒性。
4 結論
我們提出了一種新的抗癌藥物協同作用預測模型SMILESynergy。模型以藥物的SMILES作為輸入,并利用異構體數據進行數據增強,然后采用預訓練的Transformer模型編碼藥物組合數據,最后通過連接MLP完成對藥物組合協同作用的預測。在O’Neil數據集上,SMILESynergy優于其他模型,具有更好的預測性能,可幫助研究人員快速篩選最優藥物組合,提高癌癥治療效果。數據及工程文件可在https://github.com/unclestrong/SMILESynergy獲取。
重要聲明
利益沖突聲明:本文全體作者均聲明不存在利益沖突。
作者貢獻聲明:張立強主要負責實驗方案設計、代碼編寫與調試、實驗結果分析與論文撰寫,秦玉芳主要負責算法咨詢與建議、實驗結果分析、論文撰寫與指導,陳明作者為論文提供了資助和支持。
本文附件見本刊網站的電子版本(biomedeng.cn)。