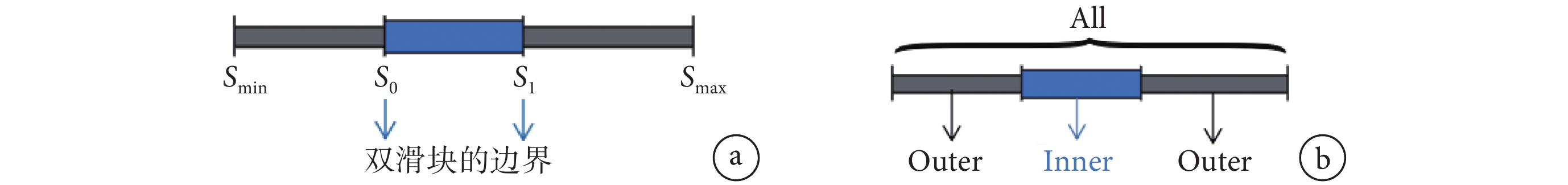人腦作為一個復雜的系統,其拓撲結構在研究腦結構和功能機制上有十分重要的作用。利用圖論可以有效地分析復雜腦網絡并且得到它在正常發育和病理狀況下拓撲結構的變化。為了更加高效地利用圖論的分析策略,開發具有圖形界面的可視化軟件非常必要。為此,我們開發了 VisConnectome,以直觀、友好的方式呈現腦網絡的分析結果。該軟件提供獨特的圖形用戶界面(GUI),包括工具箱、工具欄、雙滑塊過濾器、腦區屬性欄等,可以在任意微軟 Windows 操作系統下運行且不依賴于其他平臺。讀入自定義初始化腦網絡信息的腳本文件后,將腦網絡抽象為球棍模型并允許相關可視化操作,如標識節點和邊、改變屬性(如大小和顏色)、過濾簡化腦網絡以及與腦表面融合、腦分區顯示等。通過實驗和分析,我們得出:VisConnectome 是一個高效、快速的腦網絡可視化軟件,可以幫助研究人員更加靈活方便地對結構或功能腦網絡進行可視化和比較分析。
引用本文: 王一帆, 朱黎, 何澤睿, 楊維華, 田歌, 申佳麗, 駱巖林. VisConnectome:基于圖論且獨立運行的腦網絡可視化軟件. 生物醫學工程學雜志, 2019, 36(5): 810-817. doi: 10.7507/1001-5515.201803080 復制
版權信息: ?四川大學華西醫院華西期刊社《生物醫學工程學雜志》版權所有,未經授權不得轉載、改編
引言
人腦作為一個強大的系統,存在著復雜的拓撲結構,有許多認知疾病可以引起人腦拓撲結構的變化,如阿爾茲海默癥(Alzheimer’s disease,AD)[1]和精神分裂癥[2]等,通過其拓撲結構來揭示腦結構和功能機制是目前研究的一個重要方向。
圖論作為通用的建模方式,將復雜網絡抽象為簡單的幾何圖形,包括節點以及不同節點之間的邊[3],常用來分析人腦的拓撲屬性[4]。目前,在使用圖論分析腦網絡的拓撲關系時,大多采用的是大尺度腦網絡模型[3]。其中,節點是由大腦解剖(或功能)模板劃分的感興趣區(region of interest,ROI);連接節點的邊則主要由節點之間的結構(或功能)連接關系決定[5],結構連接關系通過白質纖維的連接情況表示[6-7],而功能連接關系主要取決于兩個節點信號的時間同步性和功能上的依賴關系[8-9]。對腦區和腦區之間的連接情況進行結構性的描述,有助于在人腦結構基礎上理解其功能。
隨著圖論在復雜腦網絡中的應用,一些具有圖形界面的腦網絡可視化軟件發展起來。Caret(http://brainvis.wustl.edu/wiki/index.php/Caret:About)是一個用于對大腦或小腦皮質進行結構和功能性研究的軟件包,既可以在圖形界面上操作和顯示神經影像數據,又可以使用命令行批量處理神經影像數據,但是它更側重于大腦皮層的可視化與分析,缺乏對腦網絡拓撲關系的研究。GAT[10]是基于 Matlab 的工具包,其圖形用戶界面(Graphical User Interface,GUI)用來比較分析結構和功能腦網絡的拓撲屬性差異。Connectome Viewer[11]是基于 Python 的可視化工具,不僅可以進行大腦分區顯示還可以顯示腦網絡拓撲結構。而 Brainnet Viewer[12]則是一個基于 Matlab 的廣泛使用的腦網絡可視化工具,可按照感興趣區對腦表皮進行分區,還可對腦網絡的球棍模型進行用戶交互操作,但其可視化速度有待提高。
為了解決上述問題,我們選擇 Microsoft Foundation Classes(MFC)作為程序框架,讀取腦節點和連接關系的信息,并通過 OpenGL 實現腦網絡的可視化,提高繪制的質量與效率。
1 相關技術
1.1 數據定義
在繪制腦網絡之前,需要導入自定義腳本文件:coord 文件、colorramp 文件、node 文件、connection 文件和 surface 文件。其中,coord 文件包含所有節點名稱及三維坐標,colorramp 文件中顏色組合用于腦網絡著色,node 文件由節點名稱及其各項屬性(如節點大小)構成,connection 文件包含具有連接關系的節點名稱以及邊的屬性(如顏色、大小),surface 文件包含不同腦區頂點和面信息。
自定義腳本文件中,有 Coord、ColorRamp 和 Window 三個模塊,如圖 1 所示。在 Coord 和 ColorRamp 模塊中,直接調用對應文件。而在模塊 Window 中,有 Node、Connection、Surface 和 Annotation 四個對象,首先引用對應的文件,然后根據定義的初始顏色和透明度給不同對象賦值,其中在初始化 Node 和 Connection 對象時,需設置大小范圍、過濾顯示范圍等。
 圖1
自定義腳本文件
Figure1.
Custom script file
圖1
自定義腳本文件
Figure1.
Custom script file
1.2 腦網絡可視化
1.2.1 數據預處理
從 Multi-Modal MRI Reproducibility Resource(http://www.nitrc.org/projects/multimodal)下載得到使用的彌散張量成像(diffusion tensor imaging,DTI)數據。原始的人腦核磁共振數據被許多不可控因素影響,因此我們使用 PANDA(http://www.nitrc.org/projects/panda/)對數據進行預處理。具體步驟如下:
① 圖像校正:利用空間位置校正去除數據掃描時被試頭部晃動帶來的噪聲,并減弱成像時渦流效應造成的誤差[13]。
② 空間配準:對被試的 T1 結構圖像進行醫學圖像配準[14],然后將配準參數作用在 DTI 圖像上,將 DTI 圖像歸入蒙特利爾神經學研究所提出的坐標系(Montreal Neurological Institute,MNI)進行標準化。
③ 腦區模塊化:利用 MNI 機構提供的 Anatomical Automatic Labeling(AAL)[15]標準模板對腦區進行模塊劃分,每個腦區可看作一個節點。
④ 平滑:采用 AAL 標準模板消除個體大腦形態結構之間差異后,利用平滑進一步消除細微差異。
1.2.2 構建腦網絡
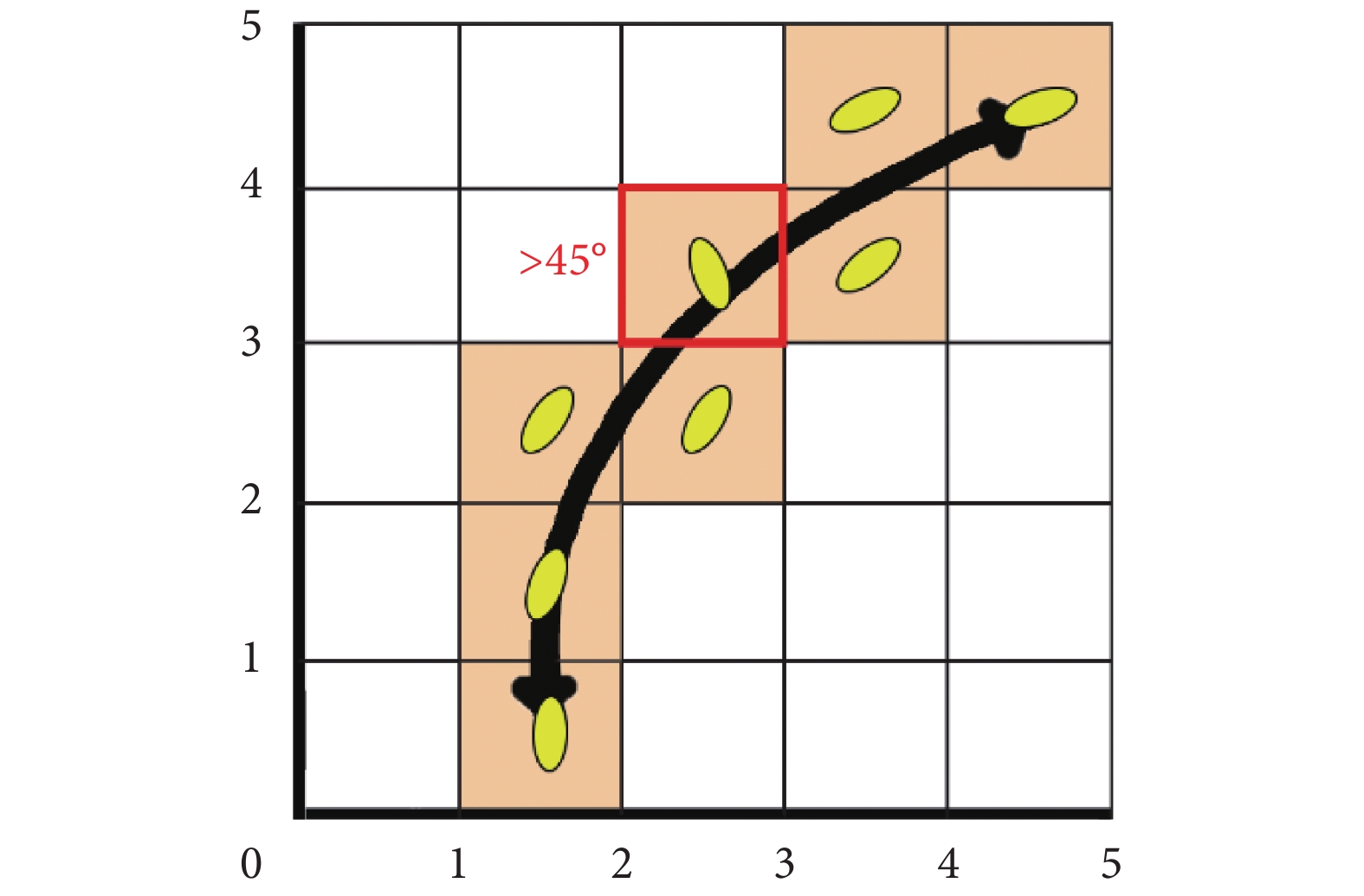
在獲取腦網絡節點后,邊由基于彌散張量成像的纖維追蹤方法獲取。追蹤過程中采用確定性追蹤算法中的連續纖維跟蹤法(fiber assignment by continuous tracking,FACT)[16]。選定隨機種子數,從中心種子開始,通過它的主向量的正、反方向分別進行延伸至體素邊界。纖維延伸過程中遇到新的體素時,當前種子是否添加到纖維中由它的主向量方向與延伸方向之間的夾角確定,如此反復直到達到追蹤的終止條件[16],如圖 2 所示,以種子點的中央(坐標是 (2.5, 2.5))作為出發點,沿各向異性程度較高的鄰域體素跟蹤,作為下一走向。在實驗中發現,臨界夾角設置過大可產生虛假纖維,過小則丟失部分目標纖維,當臨界夾角設置為 45°,構建的腦網絡完整性和準確性最高。因此,實驗設定臨界夾角為 45°,即當前體素與纖維延伸方向夾角小于 45° 時,屬于擴散纖維。
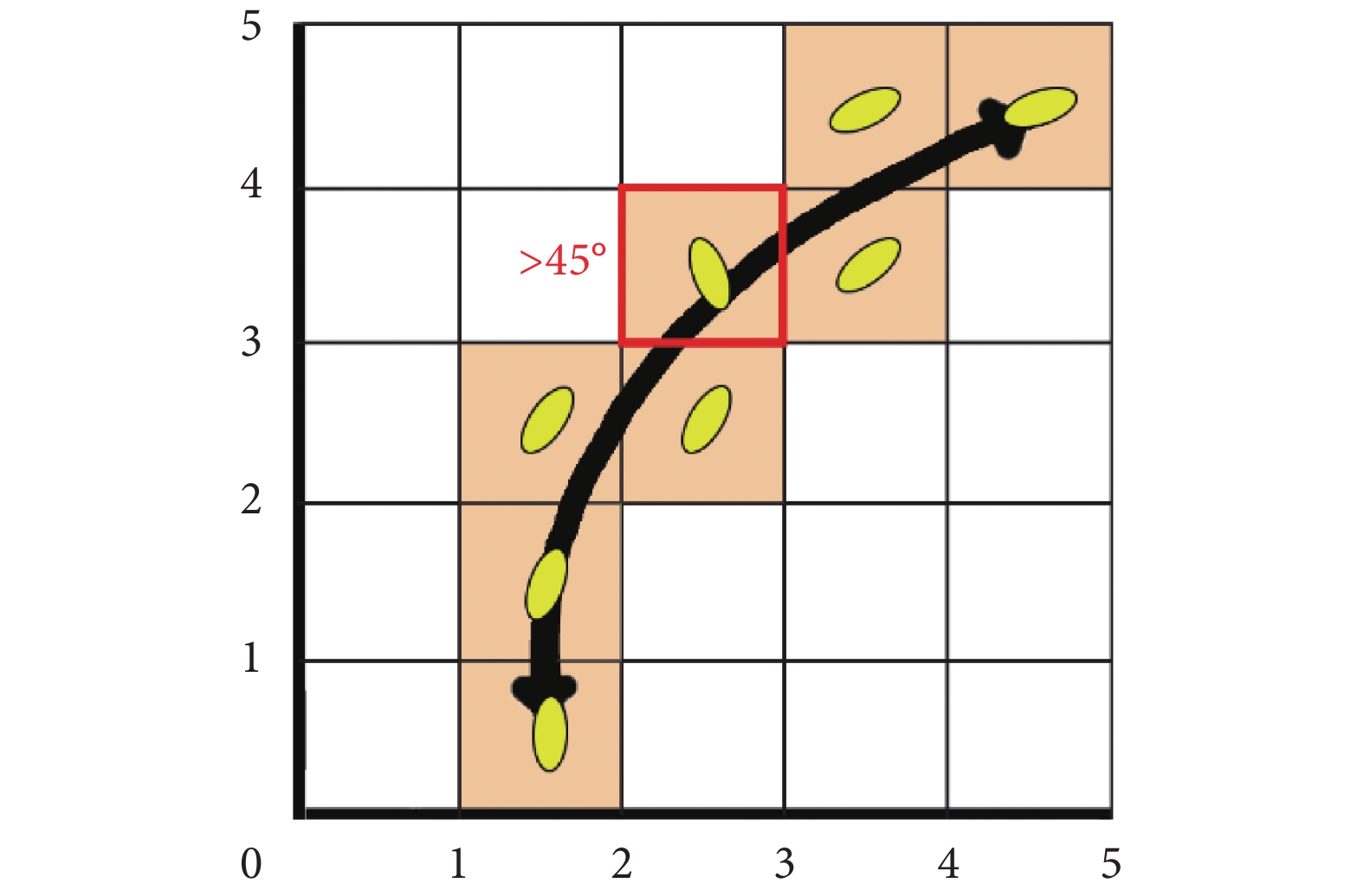 圖2
FACT算法擴散纖維延伸過程
圖2
FACT算法擴散纖維延伸過程
黃色橢圓表示體素,陰影表示該區域的體素各向異性程度較高,帶箭頭的黑色實線表示跟蹤出的纖維路徑,紅色框線表示體素的臨界夾角超過 45° 不選取該體素方向
Figure2. Diffusion fiber extension process based on FACT algorithmthe yellow ellipse represents voxel; the shadows indicate that the voxel anisotropy in this region is high; the black solid line with arrows indicates the tracked fiber path; the red frame indicates that the critical angle of voxel is more than 45 degrees, and the voxel direction is not selected
1.2.3 繪制腦網絡
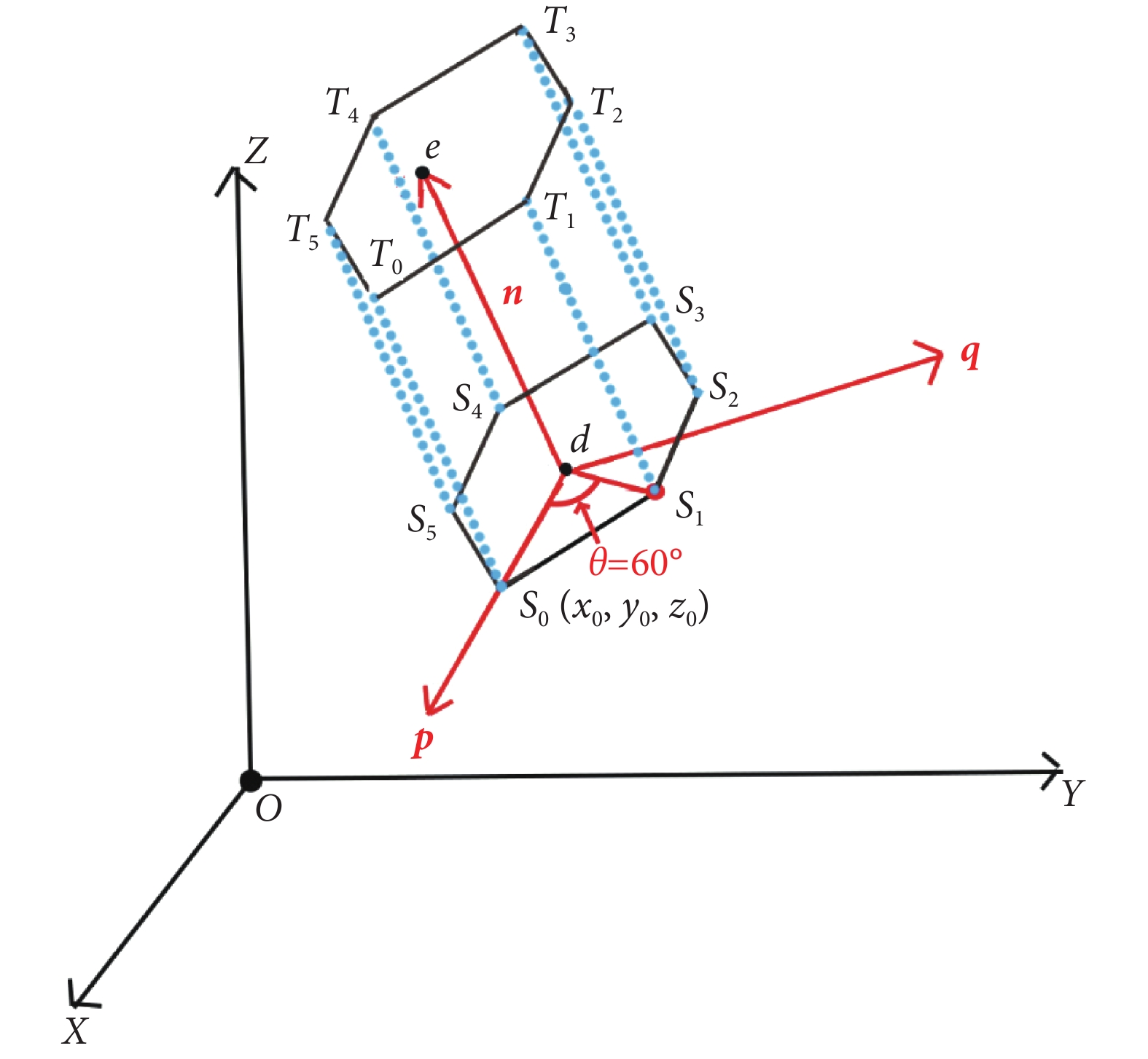
步驟 1:繪制節點和邊的幾何模型。利用球模型來表示腦節點,六面體所形成的管狀曲面表示邊,通過繪制六面體代替圓柱體來減少繪制次數并且提高可視化速度。如圖 3 所示,已知起始位置  和終止位置
和終止位置  ,以及管狀底面半徑 r 和方向向量
,以及管狀底面半徑 r 和方向向量  。對向量 n 的 Y 坐標值增加小幅度(如 0.01)變化,形成新的向量 m,由 n 與 m 形成平面P,其法向量
。對向量 n 的 Y 坐標值增加小幅度(如 0.01)變化,形成新的向量 m,由 n 與 m 形成平面P,其法向量  。定義
。定義  ,對向量
,對向量  與向量
與向量  進行單位化:
進行單位化:
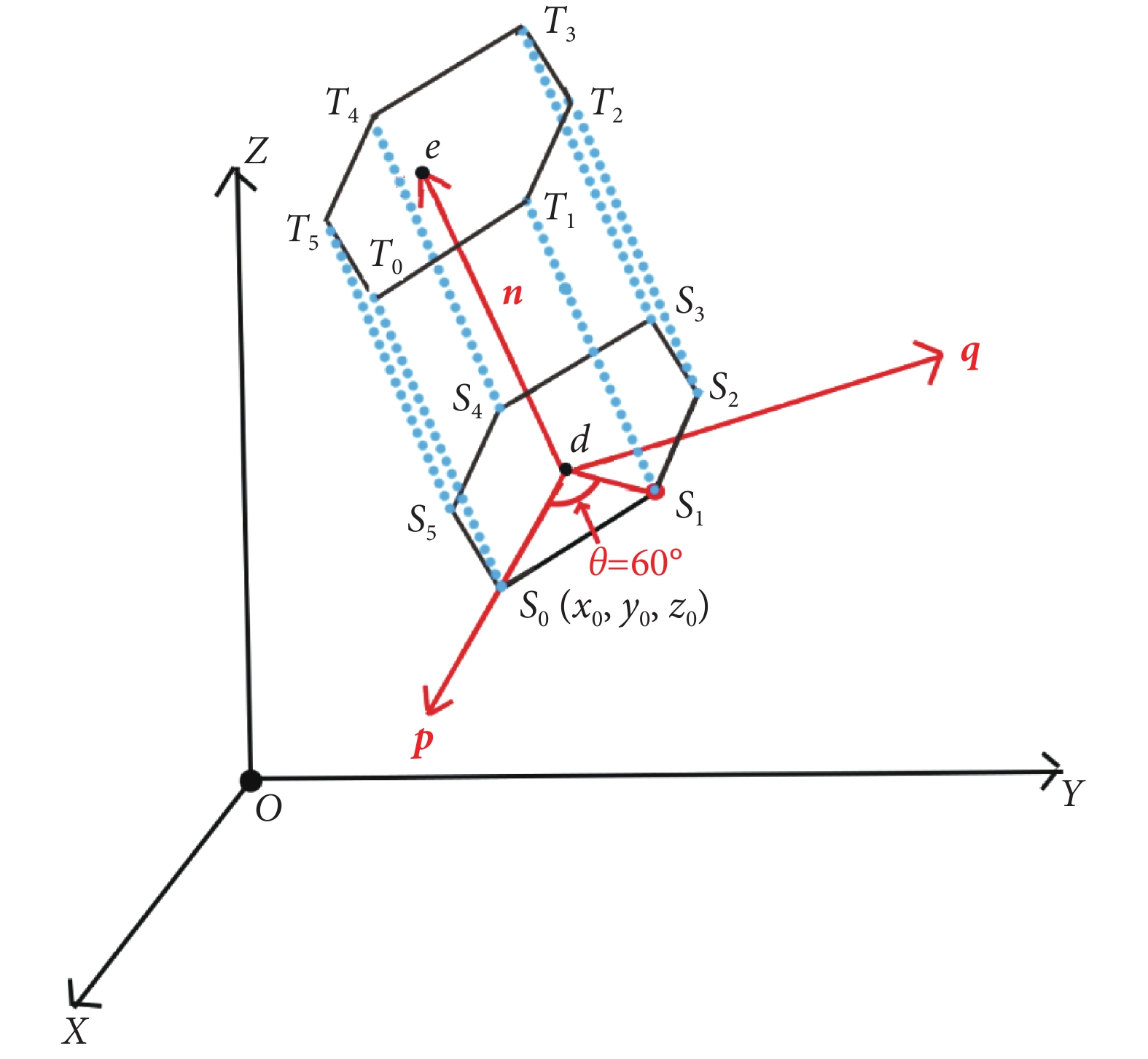 圖3
管狀曲面示意圖
Figure3.
Schematic diagram of tubular surface
圖3
管狀曲面示意圖
Figure3.
Schematic diagram of tubular surface
 |
由 p、q 形成的平面 L 垂直于 n,以  為中心,假設
為中心,假設  ,六面體的底面頂點
,六面體的底面頂點  為:
為:
 |
對管狀頂面進行相同操作形成頂點
 ,繪制每個矩形
,繪制每個矩形
 。
。
步驟 2:利用 Color Ramp 著色。根據屬性范圍(如大小)來繪制節點和邊的不同顏色。假設 colorramp 文件中定義的顏色數為N,顏色序列為 。當前要繪制的節點為M,它的大小 X 在區間[A, B]內,則它的相對位置 ξ 為:
。當前要繪制的節點為M,它的大小 X 在區間[A, B]內,則它的相對位置 ξ 為:
 |
根據 ξ,節點所對應的顏色索引為:
 |
故在顏色序列 C 中,Ci 是繪制節點 M 選取的顏色。上述著色方法同樣適用于腦網絡的邊。
步驟 3:確定節點大小。腳本文件中所引用節點屬性決定它們的大小,通過遍歷 node 文件中定義的節點屬性可得最大值 Rmax 和最小值 Rmin,Ri 是當前節點的屬性值。如圖 4a 所示,S0 和 S1 是雙滑塊的邊界,最大值和最小值為 Smax 和 Smin,我們在程序里設定 Smax 和 Smin 的值。當通過滑動雙滑塊的邊界改變當前節點的大小時,節點的實際顯示大小 Si 由它的初始大小和雙滑塊的邊界大小共同決定:
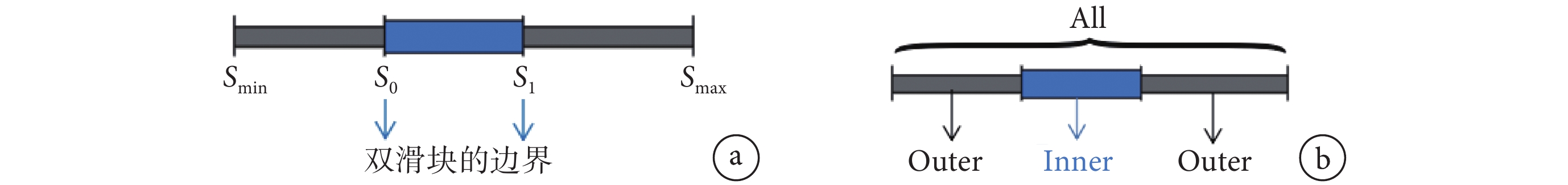 圖4
雙滑塊示意圖
圖4
雙滑塊示意圖
a. 用于調整大小;b. 過濾器
Figure4. Schematic diagram of double slidera. for resizing; b. for filtering
 |
步驟 4:管狀曲面的大小。與步驟 3 算法類似,Rmax 和 Rmin 由 connection 文件得到,Smax 和 Smin 與上面定義的相同。
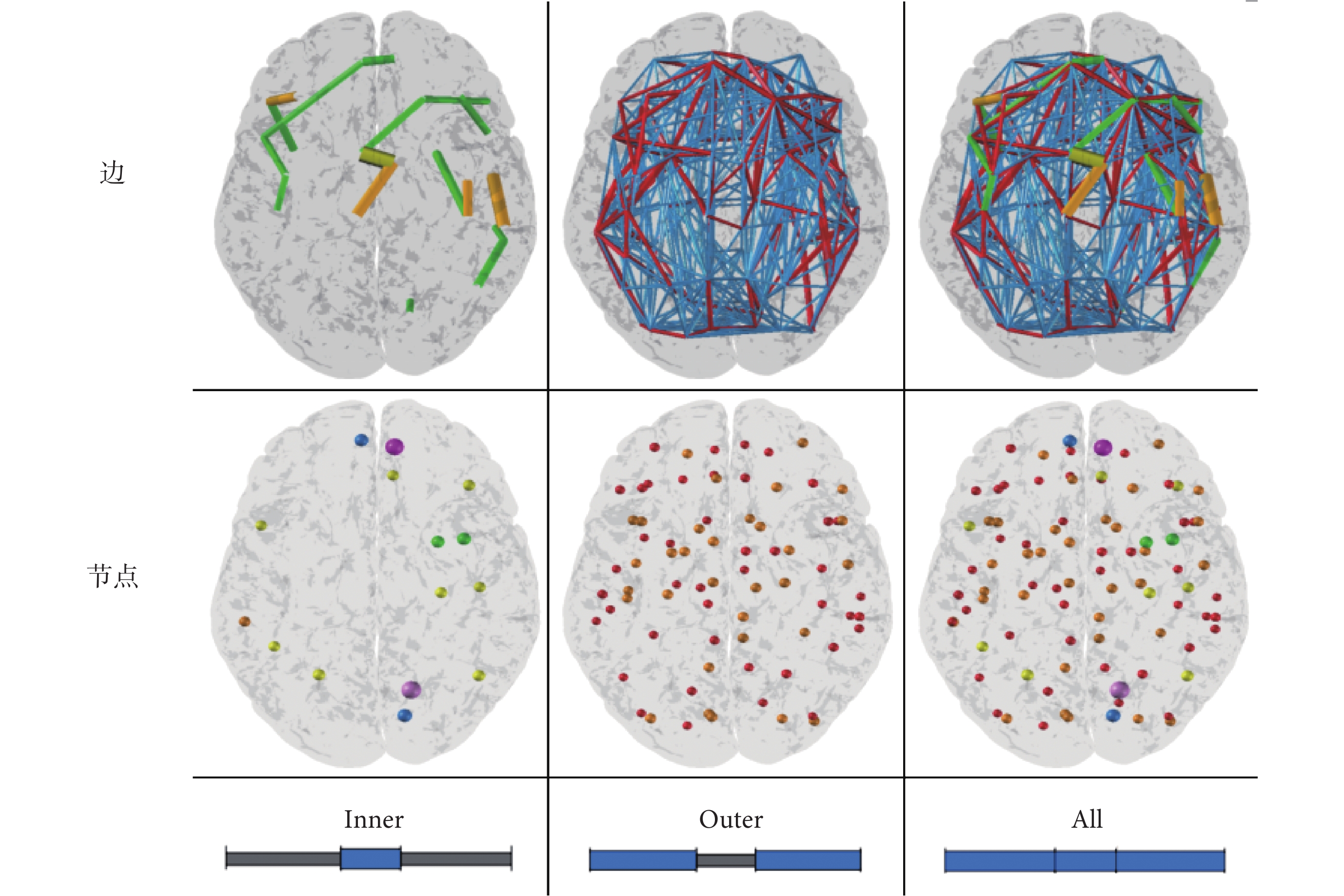
步驟 5:過濾顯示腦網絡。由于大腦具有復雜的連接關系,我們設計的過濾器用于顯示局部的腦網絡結構,圖 4b 為雙滑塊示意圖,根據節點或邊特定屬性(如大小),將腦網絡分為 Inner 和 Outer 兩部分。Inner 部分表示在雙滑塊邊界所確定的屬性值范圍內的節點或邊,而在 Outer 部分中顯示剩余的節點或邊,過濾效果如圖 5 所示。
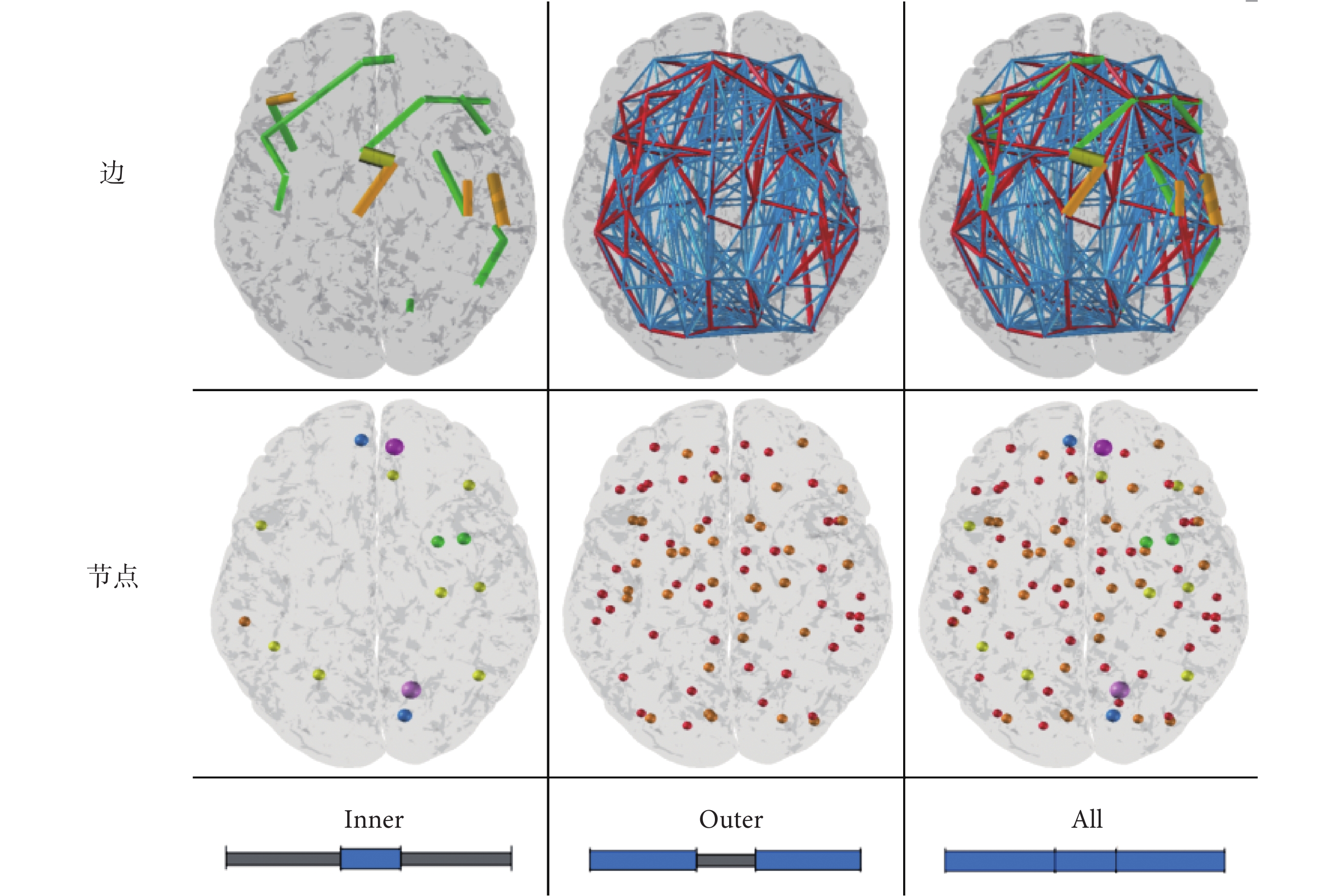 圖5
雙滑塊過濾器顯示腦網絡效果圖
Figure5.
The renderings of brain networks using double slider filter
圖5
雙滑塊過濾器顯示腦網絡效果圖
Figure5.
The renderings of brain networks using double slider filter
步驟 6:顯示腦分區。腦表皮對于腦網絡是非常重要的關聯信息,用戶可以通過腦表皮觀察節點所對應的腦區位置。在 surface 文件加載后,系統通過繪制三角面片形成腦表皮。
① 從 BrainNet Viewer(https://www.nitrc.org/frs/?group_id=504)開源數據中獲得帶有腦分區信息的 nii 文件,利用 Matlab 的 SPM12 包對數據進行降維處理,并轉為 txt 格式方便后續使用。
② 提取腦區邊緣數據。尋找邊緣點,使用處理后的數據繪制出原始的腦表皮。
③ 判斷邊緣點。由于輸入數據是一個M × N × P的三維體數據,在構成 obj 文件時首先要找到邊緣上的點。邊緣點并不好定義,但是對于內部點很好界定。在考慮單個點的情況時,位于三維體數據的中間點通常有 26 個,如圖 6 所示。對單個點情況進行推廣,通過對點周圍數據的判斷,將點分為以下三種類型,從而對三維數據進行降維處理:
 圖6
三維點矩陣模型
Figure6.
Three-dimensional point matrix model
圖6
三維點矩陣模型
Figure6.
Three-dimensional point matrix model
? 區域點:與區域值相等的點,標記該點所屬腦區;
? 內部點:在點周圍 26 個方向上的點都是同一區域點,那么這個點就是該區域的一個內部點;
? 邊緣點:在點周圍 26 個方向上存在不同區域的點。
④ 前面已經獲取了邊緣點,我們需要在此基礎上構建表面,實現腦區表面的繪制,利用邊界點集構建邊界面。利用 OpenGL 相關函數呈現腦分區,根據所有 obj 文件繪制不同腦區,并對模型隨機著色。
2 結果與分析
實驗中,腦網絡的節點由 AAL 模板劃分得到,邊的連接強度由擴散纖維上的體素個數代表,節點和大腦表皮處于同一坐標系,即 MNI 坐標系。VisConnectome 以 C++作為編程語言,在 Windows64 位操作系統(Microsoft Corp.,美國)下開發。使用 MFC 控件構成圖形界面,并調用 OpenGL 繪制腦網絡,獲得平均幀速率約 210 幀/秒。其 GUI 提供便捷的用戶交互操作,通過雙滑塊設置 Inner 和 Outer 范圍,可顯示任一部分腦網絡。而 BrainNet Viewer 只顯示高于設定閾值的部分[12]。
2.1 圖形用戶界面
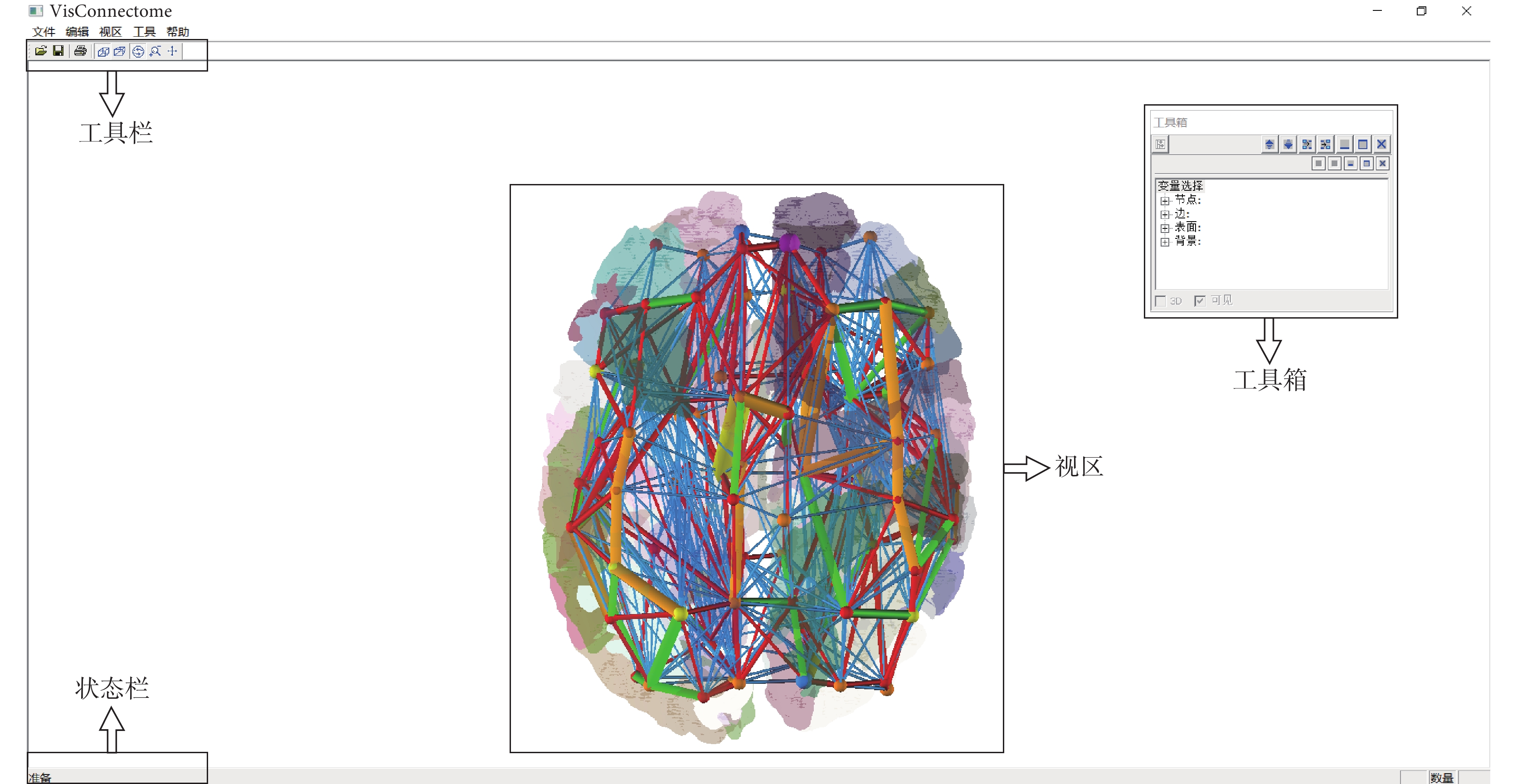
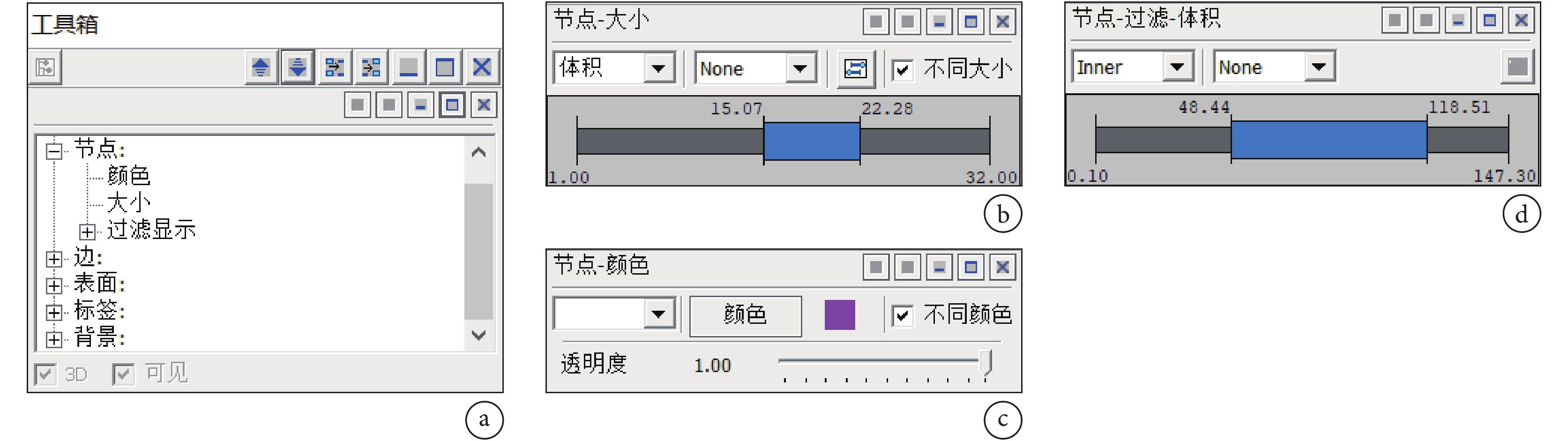
圖形界面由工具箱、工具欄、狀態欄和視區四部分組成,如圖 7 所示。工具欄按鈕對應菜單中的幾何操作,工具箱的操作界面如圖 8 所示,節點模塊中包含顏色、大小和過濾顯示,分別對應的功能為顏色及透明度調整、大小設置以及過濾顯示局部腦網絡。邊模塊所含功能與節點模塊類似。
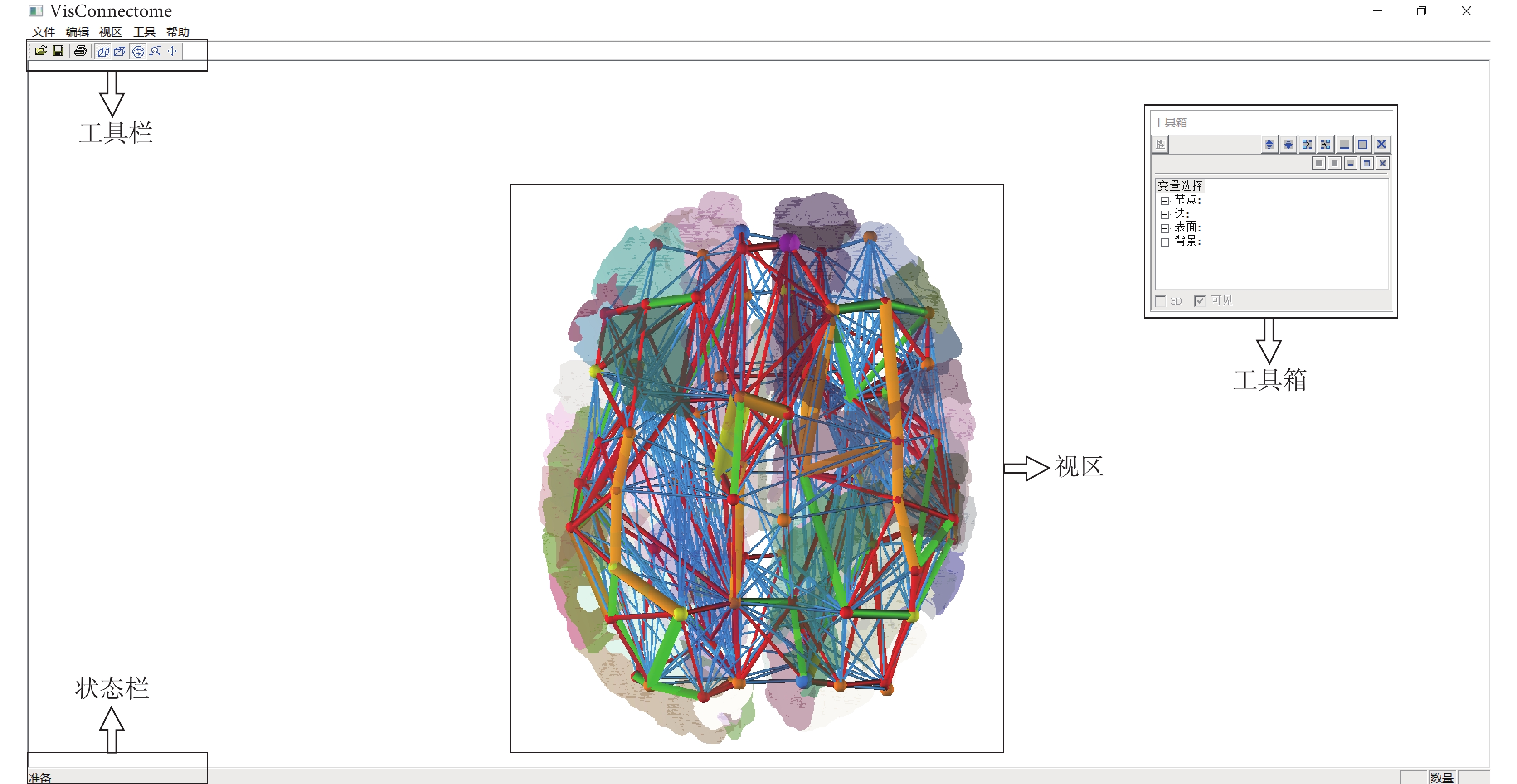 圖7
VisConnectome窗口界面
Figure7.
GUI of VisConnectome
圖7
VisConnectome窗口界面
Figure7.
GUI of VisConnectome
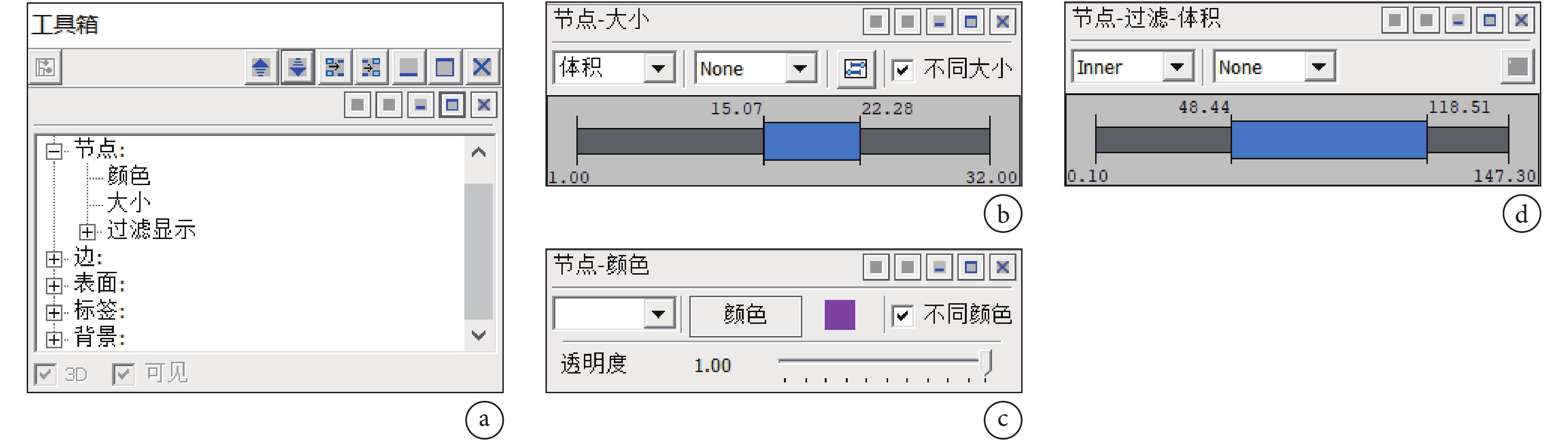 圖8
工具箱
圖8
工具箱
a. 操作對象;b. 調節大小的面板;c. 顏色調色板及透明度滑動條;d. 雙滑塊過濾器選項
Figure8. Toolboxa. toolbox objects; b. resizing panel; c. color palette and transparency slider; d. double slider filter options
2.2 腦網絡可視化效果
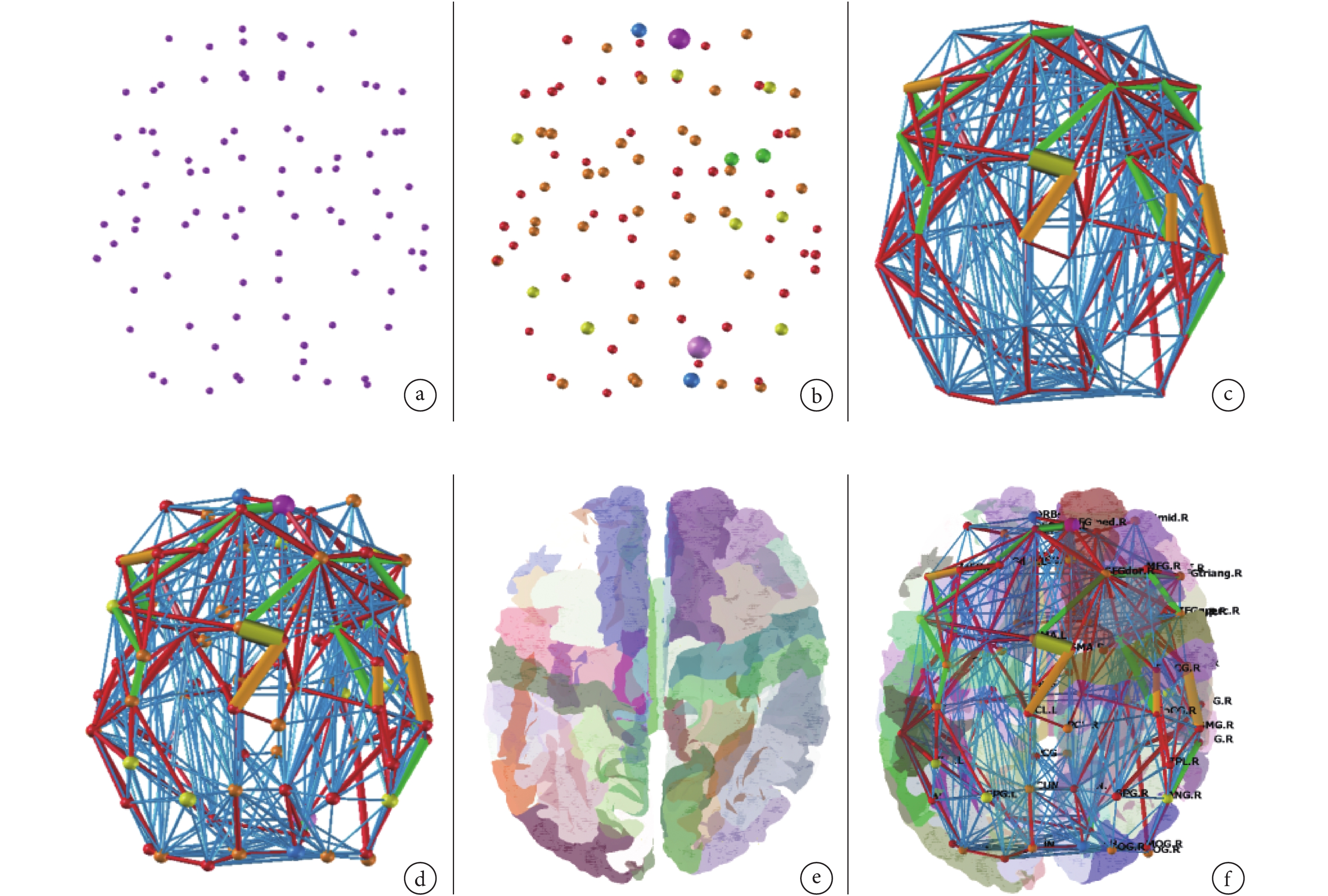
自定義腳本文件中腦網絡初始的顏色和透明度必須設置,通過關鍵字“size”“ramp”和“filter”決定大小調節、分層著色和過濾顯示功能。同時,腳本文件包含 coord、node、connection、surface 和 colorramp 等子文件信息,不同子文件組合可以寫入到腳本文件中,如圖 9 所示。腦分區結果的不同視角如圖 10 所示。
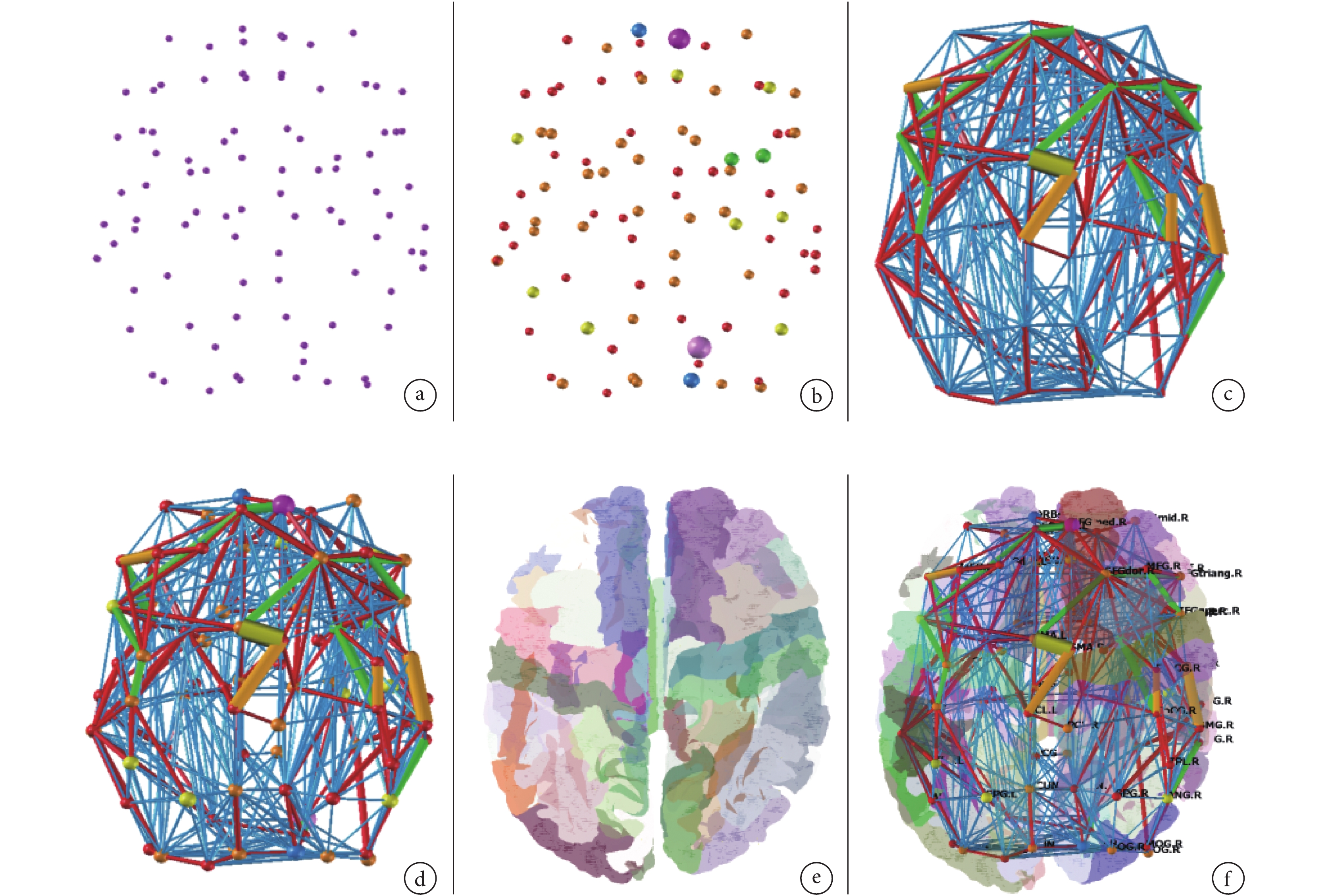 圖9
不同文件組合下的腦網絡可視化效果
圖9
不同文件組合下的腦網絡可視化效果
組合方式為:a. coord 文件;b. coord、color ramp 和 node 文件;c. coord、color ramp 和 connection 文件;d. coord、color ramp、node 和 connection 文件;e. coord、color ramp 和 surface 文件;f. coord、color ramp、node、connection、surface 和 annotation 文件
Figure9. Visualization of brain networks under different file combinationsa. coord file; b. coord, color ramp and node files; c. coord, color ramp and connection files; d. coord, color ramp, node and connection files; e. coord, color ramp and surface files; f. coord, color ramp, node, connection, surface and annotation files
 圖10
不同視角的腦分區
Figure10.
Brain regions of different perspectives
圖10
不同視角的腦分區
Figure10.
Brain regions of different perspectives
2.3 正常和病理變化的腦網絡的比較
對神經精神疾病引起分離的腦區結構和功能變異的研究已經很多,但神經精神疾病往往不止由分離的腦區結構和功能變異誘發,而是很多腦區結構功能變異共同作用的結果[17]。復雜腦網絡分析對于研究神經精神疾病的多腦區作用機制具有獨到優勢。
因此,我們選擇 ADNI 網站(https://ida.loni.usc.edu/login.jsp?project=ADNI)上項目 136_S_1227 中的 AD 患者數據(該患者患病 5 年,處于 AD 第二階段中度癡呆期,表現為遠近記憶嚴重受損,簡單結構的視空間能力下降,時間、地點定向障礙;不能獨立進行室外活動,在穿衣和個人衛生等方面需要幫助;可見失語、失用和失認)。
腦網絡類聚系數為網絡內所有節點的類聚系數 Ci 的均值,而節點 i 的類聚系數 Ci 定義為:網絡 G 內與節點 i 直接相連的“其他節點”之間的邊數與這些“其他節點”之間的最大可能邊數之間的比值,見式(6)。其中,Ei 代表與節點 i 直接相連的“其他節點”之間的邊數,Di 即與節點 i 直接相連的“其他節點”數目。
 |
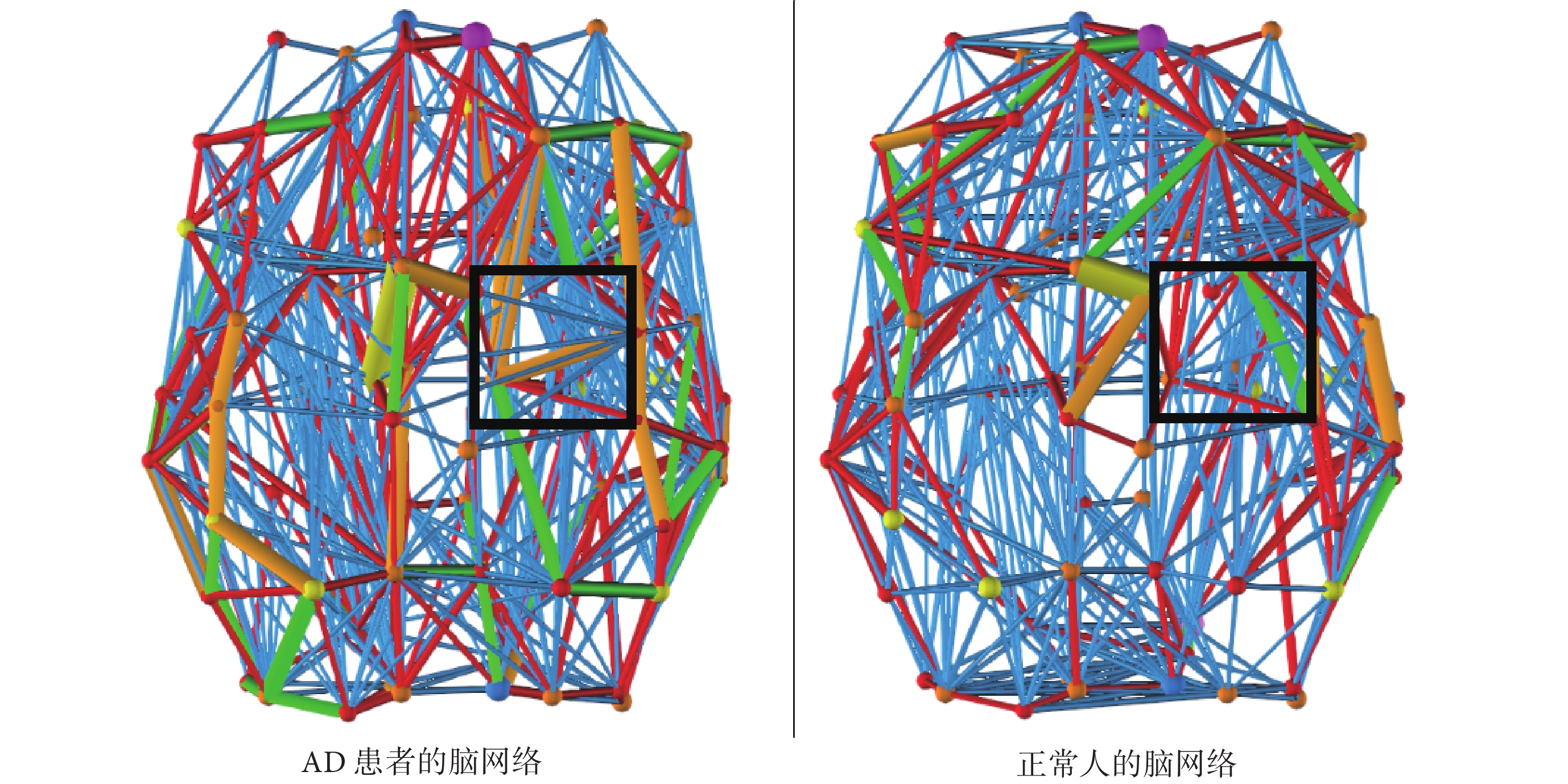
可以看出Ci表征的是節點i的相鄰節點組成的“團”的緊湊程度。按照 1.2 節的方法進行處理,并與 1.2 節所提及的正常人的腦網絡進行可視化效果比較,圖 11 直觀表現了它們之間的差異:AD 患者的腦網絡類聚系數顯著小于正常人。AD 患者腦網絡中節點所在“團”的緊湊程度大大下降,而在一些特定的區域(圖中黑框標記)節點間連接強度增強。可證明 AD 患者腦網絡拓撲結構發生了病理性的變化,并且可以以腦網絡類聚系數為依據實現對正常人和 AD 患者之間的分類,這與 Supekar 等[18]基于腦電技術(electroencephalogram,EEG)得到的 AD 患者腦結構變化相吻合。
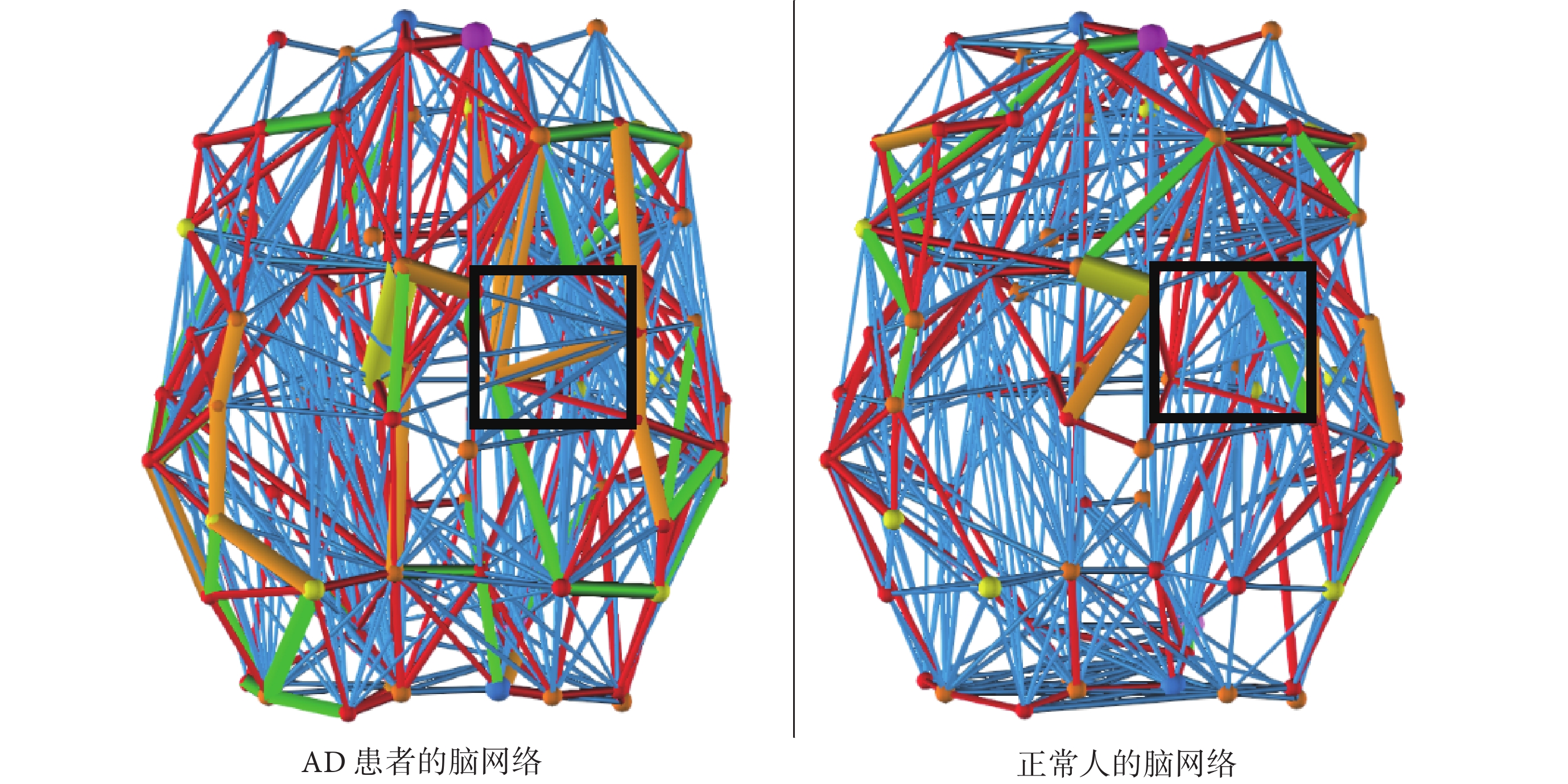 圖11
正常人和AD患者腦網絡在特定區域的可視比較分析
Figure11.
Visual comparative analysis of brain networks of normal person and AD patient in specific regions
圖11
正常人和AD患者腦網絡在特定區域的可視比較分析
Figure11.
Visual comparative analysis of brain networks of normal person and AD patient in specific regions
3 結論
基于圖論的復雜腦網絡分析已經廣泛展開,并且在正常人腦網絡特性分析和患者腦網絡變異分析中取得很多有價值的成果。但目前該領域的研究還處于起步階段,有很多問題需要結合可視化技術深入探索,比如腦結構網絡與功能網絡的相互影響關系、功能網絡拓撲結構隨時間的變化,以及網絡拓撲結構參數與認知、行為的關系等。
VisConnectome 具有創新用戶界面,如工具箱、工具欄、雙滑塊過濾器等設計,以平均 210 幀/秒速度實現快速可視化,不依賴其他平臺(如 Matlab),在 Windows 操作系統下獨立運行。利用它可以方便地進行腦網絡的系列可視化操作,如節點和邊的標識、屬性改變、過濾簡化以及與腦表面融合等。VisConnectome 是一個高效、快速的腦網絡可視化軟件,幫助研究人員更加靈活方便地對結構或功能腦網絡進行可視化,發現腦網絡異常。
VisConnectome 仍然需要在一些地方進一步改進,如未來和腦網絡領域專家合作,根據需求擴展可視分析功能,將過濾顯示和分層著色與局部腦網絡分析結合起來;此外,從繪制的速度和質量方面也有待進一步提高,如采用基于圖形處理器(graphics processing unit,GPU)的 Compute Unified Device Architecture(CUDA)框架提高速度。
利益沖突聲明:本文全體作者均聲明不存在利益沖突。
引言
人腦作為一個強大的系統,存在著復雜的拓撲結構,有許多認知疾病可以引起人腦拓撲結構的變化,如阿爾茲海默癥(Alzheimer’s disease,AD)[1]和精神分裂癥[2]等,通過其拓撲結構來揭示腦結構和功能機制是目前研究的一個重要方向。
圖論作為通用的建模方式,將復雜網絡抽象為簡單的幾何圖形,包括節點以及不同節點之間的邊[3],常用來分析人腦的拓撲屬性[4]。目前,在使用圖論分析腦網絡的拓撲關系時,大多采用的是大尺度腦網絡模型[3]。其中,節點是由大腦解剖(或功能)模板劃分的感興趣區(region of interest,ROI);連接節點的邊則主要由節點之間的結構(或功能)連接關系決定[5],結構連接關系通過白質纖維的連接情況表示[6-7],而功能連接關系主要取決于兩個節點信號的時間同步性和功能上的依賴關系[8-9]。對腦區和腦區之間的連接情況進行結構性的描述,有助于在人腦結構基礎上理解其功能。
隨著圖論在復雜腦網絡中的應用,一些具有圖形界面的腦網絡可視化軟件發展起來。Caret(http://brainvis.wustl.edu/wiki/index.php/Caret:About)是一個用于對大腦或小腦皮質進行結構和功能性研究的軟件包,既可以在圖形界面上操作和顯示神經影像數據,又可以使用命令行批量處理神經影像數據,但是它更側重于大腦皮層的可視化與分析,缺乏對腦網絡拓撲關系的研究。GAT[10]是基于 Matlab 的工具包,其圖形用戶界面(Graphical User Interface,GUI)用來比較分析結構和功能腦網絡的拓撲屬性差異。Connectome Viewer[11]是基于 Python 的可視化工具,不僅可以進行大腦分區顯示還可以顯示腦網絡拓撲結構。而 Brainnet Viewer[12]則是一個基于 Matlab 的廣泛使用的腦網絡可視化工具,可按照感興趣區對腦表皮進行分區,還可對腦網絡的球棍模型進行用戶交互操作,但其可視化速度有待提高。
為了解決上述問題,我們選擇 Microsoft Foundation Classes(MFC)作為程序框架,讀取腦節點和連接關系的信息,并通過 OpenGL 實現腦網絡的可視化,提高繪制的質量與效率。
1 相關技術
1.1 數據定義
在繪制腦網絡之前,需要導入自定義腳本文件:coord 文件、colorramp 文件、node 文件、connection 文件和 surface 文件。其中,coord 文件包含所有節點名稱及三維坐標,colorramp 文件中顏色組合用于腦網絡著色,node 文件由節點名稱及其各項屬性(如節點大小)構成,connection 文件包含具有連接關系的節點名稱以及邊的屬性(如顏色、大小),surface 文件包含不同腦區頂點和面信息。
自定義腳本文件中,有 Coord、ColorRamp 和 Window 三個模塊,如圖 1 所示。在 Coord 和 ColorRamp 模塊中,直接調用對應文件。而在模塊 Window 中,有 Node、Connection、Surface 和 Annotation 四個對象,首先引用對應的文件,然后根據定義的初始顏色和透明度給不同對象賦值,其中在初始化 Node 和 Connection 對象時,需設置大小范圍、過濾顯示范圍等。
 圖1
自定義腳本文件
Figure1.
Custom script file
圖1
自定義腳本文件
Figure1.
Custom script file
1.2 腦網絡可視化
1.2.1 數據預處理
從 Multi-Modal MRI Reproducibility Resource(http://www.nitrc.org/projects/multimodal)下載得到使用的彌散張量成像(diffusion tensor imaging,DTI)數據。原始的人腦核磁共振數據被許多不可控因素影響,因此我們使用 PANDA(http://www.nitrc.org/projects/panda/)對數據進行預處理。具體步驟如下:
① 圖像校正:利用空間位置校正去除數據掃描時被試頭部晃動帶來的噪聲,并減弱成像時渦流效應造成的誤差[13]。
② 空間配準:對被試的 T1 結構圖像進行醫學圖像配準[14],然后將配準參數作用在 DTI 圖像上,將 DTI 圖像歸入蒙特利爾神經學研究所提出的坐標系(Montreal Neurological Institute,MNI)進行標準化。
③ 腦區模塊化:利用 MNI 機構提供的 Anatomical Automatic Labeling(AAL)[15]標準模板對腦區進行模塊劃分,每個腦區可看作一個節點。
④ 平滑:采用 AAL 標準模板消除個體大腦形態結構之間差異后,利用平滑進一步消除細微差異。
1.2.2 構建腦網絡
在獲取腦網絡節點后,邊由基于彌散張量成像的纖維追蹤方法獲取。追蹤過程中采用確定性追蹤算法中的連續纖維跟蹤法(fiber assignment by continuous tracking,FACT)[16]。選定隨機種子數,從中心種子開始,通過它的主向量的正、反方向分別進行延伸至體素邊界。纖維延伸過程中遇到新的體素時,當前種子是否添加到纖維中由它的主向量方向與延伸方向之間的夾角確定,如此反復直到達到追蹤的終止條件[16],如圖 2 所示,以種子點的中央(坐標是 (2.5, 2.5))作為出發點,沿各向異性程度較高的鄰域體素跟蹤,作為下一走向。在實驗中發現,臨界夾角設置過大可產生虛假纖維,過小則丟失部分目標纖維,當臨界夾角設置為 45°,構建的腦網絡完整性和準確性最高。因此,實驗設定臨界夾角為 45°,即當前體素與纖維延伸方向夾角小于 45° 時,屬于擴散纖維。
 圖2
FACT算法擴散纖維延伸過程
圖2
FACT算法擴散纖維延伸過程
黃色橢圓表示體素,陰影表示該區域的體素各向異性程度較高,帶箭頭的黑色實線表示跟蹤出的纖維路徑,紅色框線表示體素的臨界夾角超過 45° 不選取該體素方向
Figure2. Diffusion fiber extension process based on FACT algorithmthe yellow ellipse represents voxel; the shadows indicate that the voxel anisotropy in this region is high; the black solid line with arrows indicates the tracked fiber path; the red frame indicates that the critical angle of voxel is more than 45 degrees, and the voxel direction is not selected
1.2.3 繪制腦網絡
步驟 1:繪制節點和邊的幾何模型。利用球模型來表示腦節點,六面體所形成的管狀曲面表示邊,通過繪制六面體代替圓柱體來減少繪制次數并且提高可視化速度。如圖 3 所示,已知起始位置  和終止位置
和終止位置  ,以及管狀底面半徑 r 和方向向量
,以及管狀底面半徑 r 和方向向量  。對向量 n 的 Y 坐標值增加小幅度(如 0.01)變化,形成新的向量 m,由 n 與 m 形成平面P,其法向量
。對向量 n 的 Y 坐標值增加小幅度(如 0.01)變化,形成新的向量 m,由 n 與 m 形成平面P,其法向量  。定義
。定義  ,對向量
,對向量  與向量
與向量  進行單位化:
進行單位化:
 圖3
管狀曲面示意圖
Figure3.
Schematic diagram of tubular surface
圖3
管狀曲面示意圖
Figure3.
Schematic diagram of tubular surface
 |
由 p、q 形成的平面 L 垂直于 n,以  為中心,假設
為中心,假設  ,六面體的底面頂點
,六面體的底面頂點  為:
為:
 |
對管狀頂面進行相同操作形成頂點
 ,繪制每個矩形
,繪制每個矩形
 。
。
步驟 2:利用 Color Ramp 著色。根據屬性范圍(如大小)來繪制節點和邊的不同顏色。假設 colorramp 文件中定義的顏色數為N,顏色序列為 。當前要繪制的節點為M,它的大小 X 在區間[A, B]內,則它的相對位置 ξ 為:
。當前要繪制的節點為M,它的大小 X 在區間[A, B]內,則它的相對位置 ξ 為:
 |
根據 ξ,節點所對應的顏色索引為:
 |
故在顏色序列 C 中,Ci 是繪制節點 M 選取的顏色。上述著色方法同樣適用于腦網絡的邊。
步驟 3:確定節點大小。腳本文件中所引用節點屬性決定它們的大小,通過遍歷 node 文件中定義的節點屬性可得最大值 Rmax 和最小值 Rmin,Ri 是當前節點的屬性值。如圖 4a 所示,S0 和 S1 是雙滑塊的邊界,最大值和最小值為 Smax 和 Smin,我們在程序里設定 Smax 和 Smin 的值。當通過滑動雙滑塊的邊界改變當前節點的大小時,節點的實際顯示大小 Si 由它的初始大小和雙滑塊的邊界大小共同決定:
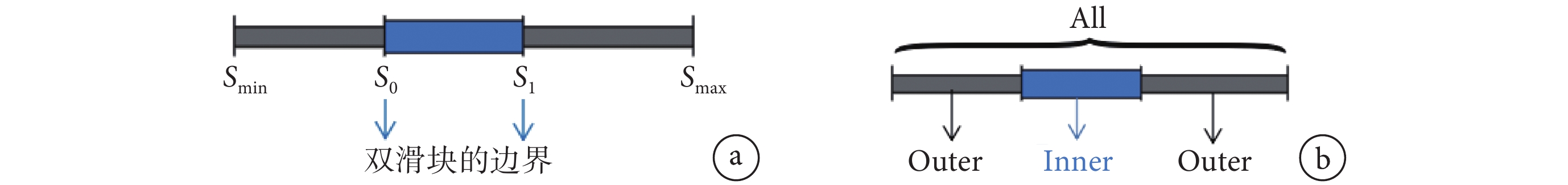 圖4
雙滑塊示意圖
圖4
雙滑塊示意圖
a. 用于調整大小;b. 過濾器
Figure4. Schematic diagram of double slidera. for resizing; b. for filtering
 |
步驟 4:管狀曲面的大小。與步驟 3 算法類似,Rmax 和 Rmin 由 connection 文件得到,Smax 和 Smin 與上面定義的相同。
步驟 5:過濾顯示腦網絡。由于大腦具有復雜的連接關系,我們設計的過濾器用于顯示局部的腦網絡結構,圖 4b 為雙滑塊示意圖,根據節點或邊特定屬性(如大小),將腦網絡分為 Inner 和 Outer 兩部分。Inner 部分表示在雙滑塊邊界所確定的屬性值范圍內的節點或邊,而在 Outer 部分中顯示剩余的節點或邊,過濾效果如圖 5 所示。
 圖5
雙滑塊過濾器顯示腦網絡效果圖
Figure5.
The renderings of brain networks using double slider filter
圖5
雙滑塊過濾器顯示腦網絡效果圖
Figure5.
The renderings of brain networks using double slider filter
步驟 6:顯示腦分區。腦表皮對于腦網絡是非常重要的關聯信息,用戶可以通過腦表皮觀察節點所對應的腦區位置。在 surface 文件加載后,系統通過繪制三角面片形成腦表皮。
① 從 BrainNet Viewer(https://www.nitrc.org/frs/?group_id=504)開源數據中獲得帶有腦分區信息的 nii 文件,利用 Matlab 的 SPM12 包對數據進行降維處理,并轉為 txt 格式方便后續使用。
② 提取腦區邊緣數據。尋找邊緣點,使用處理后的數據繪制出原始的腦表皮。
③ 判斷邊緣點。由于輸入數據是一個M × N × P的三維體數據,在構成 obj 文件時首先要找到邊緣上的點。邊緣點并不好定義,但是對于內部點很好界定。在考慮單個點的情況時,位于三維體數據的中間點通常有 26 個,如圖 6 所示。對單個點情況進行推廣,通過對點周圍數據的判斷,將點分為以下三種類型,從而對三維數據進行降維處理:
 圖6
三維點矩陣模型
Figure6.
Three-dimensional point matrix model
圖6
三維點矩陣模型
Figure6.
Three-dimensional point matrix model
? 區域點:與區域值相等的點,標記該點所屬腦區;
? 內部點:在點周圍 26 個方向上的點都是同一區域點,那么這個點就是該區域的一個內部點;
? 邊緣點:在點周圍 26 個方向上存在不同區域的點。
④ 前面已經獲取了邊緣點,我們需要在此基礎上構建表面,實現腦區表面的繪制,利用邊界點集構建邊界面。利用 OpenGL 相關函數呈現腦分區,根據所有 obj 文件繪制不同腦區,并對模型隨機著色。
2 結果與分析
實驗中,腦網絡的節點由 AAL 模板劃分得到,邊的連接強度由擴散纖維上的體素個數代表,節點和大腦表皮處于同一坐標系,即 MNI 坐標系。VisConnectome 以 C++作為編程語言,在 Windows64 位操作系統(Microsoft Corp.,美國)下開發。使用 MFC 控件構成圖形界面,并調用 OpenGL 繪制腦網絡,獲得平均幀速率約 210 幀/秒。其 GUI 提供便捷的用戶交互操作,通過雙滑塊設置 Inner 和 Outer 范圍,可顯示任一部分腦網絡。而 BrainNet Viewer 只顯示高于設定閾值的部分[12]。
2.1 圖形用戶界面
圖形界面由工具箱、工具欄、狀態欄和視區四部分組成,如圖 7 所示。工具欄按鈕對應菜單中的幾何操作,工具箱的操作界面如圖 8 所示,節點模塊中包含顏色、大小和過濾顯示,分別對應的功能為顏色及透明度調整、大小設置以及過濾顯示局部腦網絡。邊模塊所含功能與節點模塊類似。
 圖7
VisConnectome窗口界面
Figure7.
GUI of VisConnectome
圖7
VisConnectome窗口界面
Figure7.
GUI of VisConnectome
 圖8
工具箱
圖8
工具箱
a. 操作對象;b. 調節大小的面板;c. 顏色調色板及透明度滑動條;d. 雙滑塊過濾器選項
Figure8. Toolboxa. toolbox objects; b. resizing panel; c. color palette and transparency slider; d. double slider filter options
2.2 腦網絡可視化效果
自定義腳本文件中腦網絡初始的顏色和透明度必須設置,通過關鍵字“size”“ramp”和“filter”決定大小調節、分層著色和過濾顯示功能。同時,腳本文件包含 coord、node、connection、surface 和 colorramp 等子文件信息,不同子文件組合可以寫入到腳本文件中,如圖 9 所示。腦分區結果的不同視角如圖 10 所示。
 圖9
不同文件組合下的腦網絡可視化效果
圖9
不同文件組合下的腦網絡可視化效果
組合方式為:a. coord 文件;b. coord、color ramp 和 node 文件;c. coord、color ramp 和 connection 文件;d. coord、color ramp、node 和 connection 文件;e. coord、color ramp 和 surface 文件;f. coord、color ramp、node、connection、surface 和 annotation 文件
Figure9. Visualization of brain networks under different file combinationsa. coord file; b. coord, color ramp and node files; c. coord, color ramp and connection files; d. coord, color ramp, node and connection files; e. coord, color ramp and surface files; f. coord, color ramp, node, connection, surface and annotation files
 圖10
不同視角的腦分區
Figure10.
Brain regions of different perspectives
圖10
不同視角的腦分區
Figure10.
Brain regions of different perspectives
2.3 正常和病理變化的腦網絡的比較
對神經精神疾病引起分離的腦區結構和功能變異的研究已經很多,但神經精神疾病往往不止由分離的腦區結構和功能變異誘發,而是很多腦區結構功能變異共同作用的結果[17]。復雜腦網絡分析對于研究神經精神疾病的多腦區作用機制具有獨到優勢。
因此,我們選擇 ADNI 網站(https://ida.loni.usc.edu/login.jsp?project=ADNI)上項目 136_S_1227 中的 AD 患者數據(該患者患病 5 年,處于 AD 第二階段中度癡呆期,表現為遠近記憶嚴重受損,簡單結構的視空間能力下降,時間、地點定向障礙;不能獨立進行室外活動,在穿衣和個人衛生等方面需要幫助;可見失語、失用和失認)。
腦網絡類聚系數為網絡內所有節點的類聚系數 Ci 的均值,而節點 i 的類聚系數 Ci 定義為:網絡 G 內與節點 i 直接相連的“其他節點”之間的邊數與這些“其他節點”之間的最大可能邊數之間的比值,見式(6)。其中,Ei 代表與節點 i 直接相連的“其他節點”之間的邊數,Di 即與節點 i 直接相連的“其他節點”數目。
 |
可以看出Ci表征的是節點i的相鄰節點組成的“團”的緊湊程度。按照 1.2 節的方法進行處理,并與 1.2 節所提及的正常人的腦網絡進行可視化效果比較,圖 11 直觀表現了它們之間的差異:AD 患者的腦網絡類聚系數顯著小于正常人。AD 患者腦網絡中節點所在“團”的緊湊程度大大下降,而在一些特定的區域(圖中黑框標記)節點間連接強度增強。可證明 AD 患者腦網絡拓撲結構發生了病理性的變化,并且可以以腦網絡類聚系數為依據實現對正常人和 AD 患者之間的分類,這與 Supekar 等[18]基于腦電技術(electroencephalogram,EEG)得到的 AD 患者腦結構變化相吻合。
 圖11
正常人和AD患者腦網絡在特定區域的可視比較分析
Figure11.
Visual comparative analysis of brain networks of normal person and AD patient in specific regions
圖11
正常人和AD患者腦網絡在特定區域的可視比較分析
Figure11.
Visual comparative analysis of brain networks of normal person and AD patient in specific regions
3 結論
基于圖論的復雜腦網絡分析已經廣泛展開,并且在正常人腦網絡特性分析和患者腦網絡變異分析中取得很多有價值的成果。但目前該領域的研究還處于起步階段,有很多問題需要結合可視化技術深入探索,比如腦結構網絡與功能網絡的相互影響關系、功能網絡拓撲結構隨時間的變化,以及網絡拓撲結構參數與認知、行為的關系等。
VisConnectome 具有創新用戶界面,如工具箱、工具欄、雙滑塊過濾器等設計,以平均 210 幀/秒速度實現快速可視化,不依賴其他平臺(如 Matlab),在 Windows 操作系統下獨立運行。利用它可以方便地進行腦網絡的系列可視化操作,如節點和邊的標識、屬性改變、過濾簡化以及與腦表面融合等。VisConnectome 是一個高效、快速的腦網絡可視化軟件,幫助研究人員更加靈活方便地對結構或功能腦網絡進行可視化,發現腦網絡異常。
VisConnectome 仍然需要在一些地方進一步改進,如未來和腦網絡領域專家合作,根據需求擴展可視分析功能,將過濾顯示和分層著色與局部腦網絡分析結合起來;此外,從繪制的速度和質量方面也有待進一步提高,如采用基于圖形處理器(graphics processing unit,GPU)的 Compute Unified Device Architecture(CUDA)框架提高速度。
利益沖突聲明:本文全體作者均聲明不存在利益沖突。