肺癌是對人類健康威脅最大的惡性腫瘤之一。已有研究表明,一些基因在肺癌的發生發展過程中發揮著重要的調控作用。本文提出一種基于LightGBM的集成學習方法,根據免疫相關基因(IRG)表達譜數據和臨床數據構建預后模型,對肺腺癌患者的預后生存率進行預測。首先,使用Limma包進行基因差異分析,然后利用CoxPH回歸分析方法對與預后相關的IRG進行篩選,進而使用XGBoost算法對IRG特征進行重要性打分,最后利用LASSO回歸分析方法篩選可用于構建預后模型的IRG,最終結果共得到17個可用于構建模型的IRG特征。根據篩選得到的IRG特征來訓練LightGBM,使用K-means算法將患者分為三組,其模型輸出結果的受試者操作特征(ROC)曲線下面積(AUC)顯示模型預測三組患者生存率的準確率分別為96%、98%、96%。實驗結果表明,本文所提模型能夠將肺腺癌患者分為三組[5年生存率高于65%(第一組)、低于65%但高于30%(第二組)、低于30%(第三組)],并能較準確地預測肺腺癌患者的五年生存率。
引用本文: 孟祥福, 田友發, 張霄雁. 基于LightGBM模型的肺腺癌免疫相關基因篩選與患者生存率預測. 生物醫學工程學雜志, 2024, 41(1): 70-79. doi: 10.7507/1001-5515.202305038 復制
版權信息: ?四川大學華西醫院華西期刊社《生物醫學工程學雜志》版權所有,未經授權不得轉載、改編
0 引言
肺癌是世界上對人類生命健康威脅最大的惡性腫瘤之一,其發病率和死亡率已分別占到癌癥總發病率和總死亡率的11.6%和18.4%[1]。據2020年世界衛生組織國際癌癥研究機構發布的最新數據顯示,中國2020年新發肺癌83萬例,死亡人數71萬,成為世界第一肺癌大國[2-3]。此外,非小細胞肺癌(non-small cell lung cancer,NSCLC)占所有肺癌的85%以上,其中肺腺癌(lung adenocarcinoma,LUAD)的發生率最高,約占所有NSCLC的50%[4-5]。LUAD早期癥狀不明顯,確診時常處于晚期,從而錯過最佳手術時間,導致LUAD患者五年生存率一直較低,僅為15%左右;但也有數據顯示,在早期被診斷出LUAD的患者通過積極治療,其五年生存率接近100%[6-7]。
目前,LUAD臨床預后的判斷方法主要是美國癌癥聯合會(American Joint Committee on Cancer,AJCC)提出的TNM(tumor node metastasis)分期系統[8]。TNM分期系統使用三個值來描述癌癥的進展:T表示腫瘤的大小,N表示轉移至淋巴結,M表示轉移至遠處器官,較高的數字代表癌癥的更晚期。但該分期系統只考慮了腫瘤的解剖因素,而沒有考慮非解剖因素,所以不能準確地預測LUAD的預后[9]。
免疫相關基因(immune related gene,IRG)在LUAD的發生發展過程中發揮著重要的調控作用,探索異常表達的IRG并構建預后生物標志物,一方面有利于對LUAD發病機制的理解,另一方面對制定個體化診療方案以及發現新的治療靶標等具有重要意義[10]。在傳統的生存分析研究中,Cox比例風險模型(Cox proportional hazard model,CoxPH)是最常用的半參數回歸模型,其基本思想是根據患者性別、吸煙年限和身體質量指數(body mass index,BMI)等結構化特征進行生存分析。雖然回歸模型的每個變量易于解釋,但表達協變量之間的非線性關系是有限的,且無法學習圖像數據等非結構化數據。因此,集成學習和深度學習方法常用來克服線性模型的缺點[11-13]。在深度學習方面,Anika等[14]構建了基于多模態神經網絡的模型,利用臨床數據和基因表達譜數據預測20種不同癌癥類型患者的生存率。Zhu等[15]提出一種使用病理圖像進行生存分析的深度卷積神經網絡學習圖像深層次的信息,從而提高生存預測性能。在集成學習方面,Thedinga等[16]引入XGBoost(eXtreme Gradient Boosting)模型,對來自25種不同癌癥類型的8 024名患者的轉錄組數據進行患者生存率預測。
轉錄組學技術(微陣列、RNA測序)提供了關于患者的大量分子特征信息,對這些分子特征數據進行分析,進而預測癌癥患者生存率是生物醫學研究的一個重要內容[17]。在LUAD的發生發展過程中,癌細胞的變化會破壞基因的表達,從而留下有價值的信息,這些信息是多種基因表達的非線性組合。
本研究的目的是構建一種新型的基于分子特征的集成學習LUAD患者生存率預測模型。選擇IRG作為特征,分別使用Limma包、CoxPH、最小絕對收縮和選擇算子(least absolute selection and shrinkage operator,LASSO)、K-means聚類、XGBoost、LightGBM(Light Gradient Boosting Machine)和反向傳播神經網絡(back propagation neural network,BPNN)等算法進行生存相關IRG特征的識別、IRG特征的目標分層和預測模型的構建[18-25],以期提高LUAD預后預測模型的準確性,進而輔助醫生為患者提供個體化治療方案。
1 材料和方法
1.1 材料
本文使用的數據來源于癌癥基因組圖譜(The Cancer Genome Atlas,TCGA)。該數據庫是由美國國家癌癥研究所和美國國家人類基因組研究所于2006年聯合啟動建立的項目,旨在應用高通量的基因組分析技術,幫助人們對癌癥有更好的認知,從而提高對癌癥的預防、診斷和治療能力[2]。TCGA收錄了各種人類癌癥的組學數據,包括基因表達數據、拷貝數變異和DNA甲基化等。該數據庫也是癌癥研究者很重要的數據來源,目前已收錄了11 000多例患者和33種癌癥的數據。
本文采用Python整合從TCGA數據庫(https://portal.gdc.cancer.gov)下載的RNA-seq數據和臨床數據。從RNA-seq數據中獲取gene_id、gene_name和unstranded列數據來構成IRG表達數據,獲得的IRG表達數據包含510例樣本(45例正常樣本、450例肺腺癌樣本和15例轉移組樣本)和60 660個轉錄組測序結果。臨床數據是兩個Json文件,從中提取出樣本的生存時間、生存狀態、性別、AJCC分期、TNM分期((T、N、M)等數據。
1.2 方法
本文提出的方法分為3步:首先對IRG表達譜數據和臨床數據進行預處理,得到與LUAD診斷相關的IRG;然后利用K-means聚類算法對LUAD患者進行分層;最后構建基于LightGBM的肺腺癌預測模型并根據第一步得到的IRG數據和第二步的患者分層數據,對患者的生存率進行預測。源代碼在GitHub中提供(https://github.com/tianyoufa/tianyoufaCode)。
1.2.1 數據預處理
從數據中移除15例轉移組樣本,采用K最近鄰(K-nearest neighbors,KNN)算法填充缺失值,與從IMMPORT數據庫下載的免疫基因取交集,提取出與肺腺癌相關的差異表達免疫基因共2 287個。剔除在癌癥組與正常組中IRG表達量均值之比在區間[0.8, 1.2]內的IRG數據,因為該區間內的IRG在患病前與患病后無明顯變化,說明該IRG對是否患LUAD沒有影響。將臨床數據與IRG表達譜數據整合,更新患者的診斷數據,移除重復出現的患者數據,使用最新的檢測結果代替。經過預處理后的414例LUAD患者臨床數據如表1所示。
1.2.2 篩選差異表達的IRG
基于R語言及Limma包,使用log2轉化后的IRG表達譜數據對45例正常組織和414例LUAD組織進行基因差異表達分析,差異表達基因的選擇條件為fdr < 0.05且|logFC| > 2。篩選出差異基因共647個,包括上調基因348個,下調基因299個。
1.2.3 與預后相關的IRG篩選方法
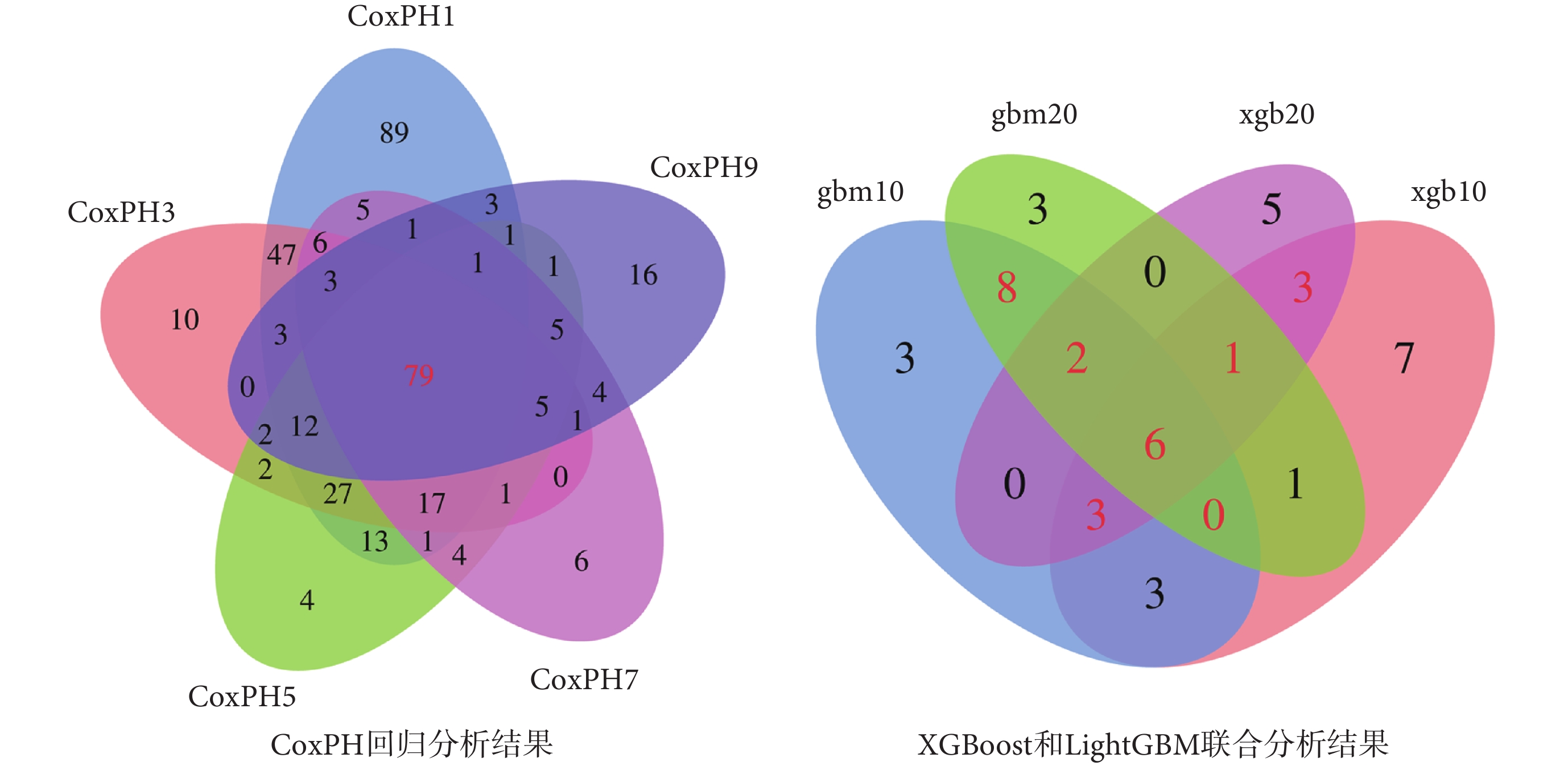
由于樣本量較少而基因較多,導致在高維空間中數據存在一定冗余信息和噪聲信息,這些信息會在建模過程中引入誤差,而對數據降維可降低模型的復雜度并提高模型的精度。因此,在得到差異表達分析結果后,先對差異表達基因以P < 0.1為閾值進行單因素CoxPH回歸分析,對數據進行降維,再以P < 0.05為閾值進行多因素CoxPH回歸分析,并對實驗結果取交集得到與預后相關的79個IRG。將CoxPH回歸分析得到的IRG輸入XGBoost與LightGBM模型進行實驗分析,對IRG的分析結果進行打分,再對XGBoost的兩組實驗結果取交集,對LightGBM的兩組實驗結果取交集,最后將XGBoost與LightGBM的實驗結果取并集,最終得到23個與預后顯著相關的IRG(如圖1所示)。
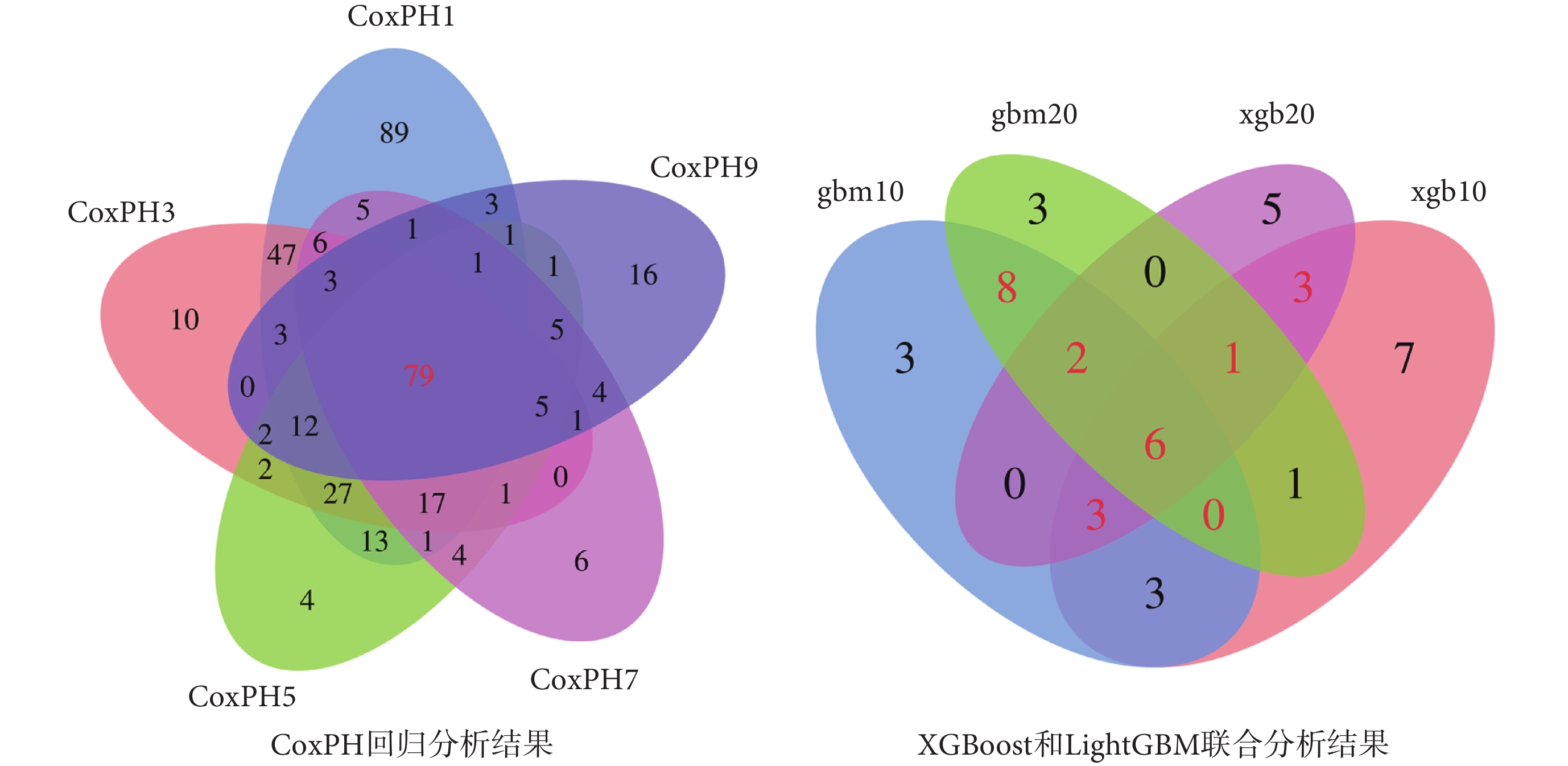 圖1
多因素Cox回歸分析與XGBoost和LightGBM聯合分析結果的韋恩圖
Figure1.
Venn diagram of multivariate Cox regression analysis and joint analysis of XGBoost and LightGBM
圖1
多因素Cox回歸分析與XGBoost和LightGBM聯合分析結果的韋恩圖
Figure1.
Venn diagram of multivariate Cox regression analysis and joint analysis of XGBoost and LightGBM
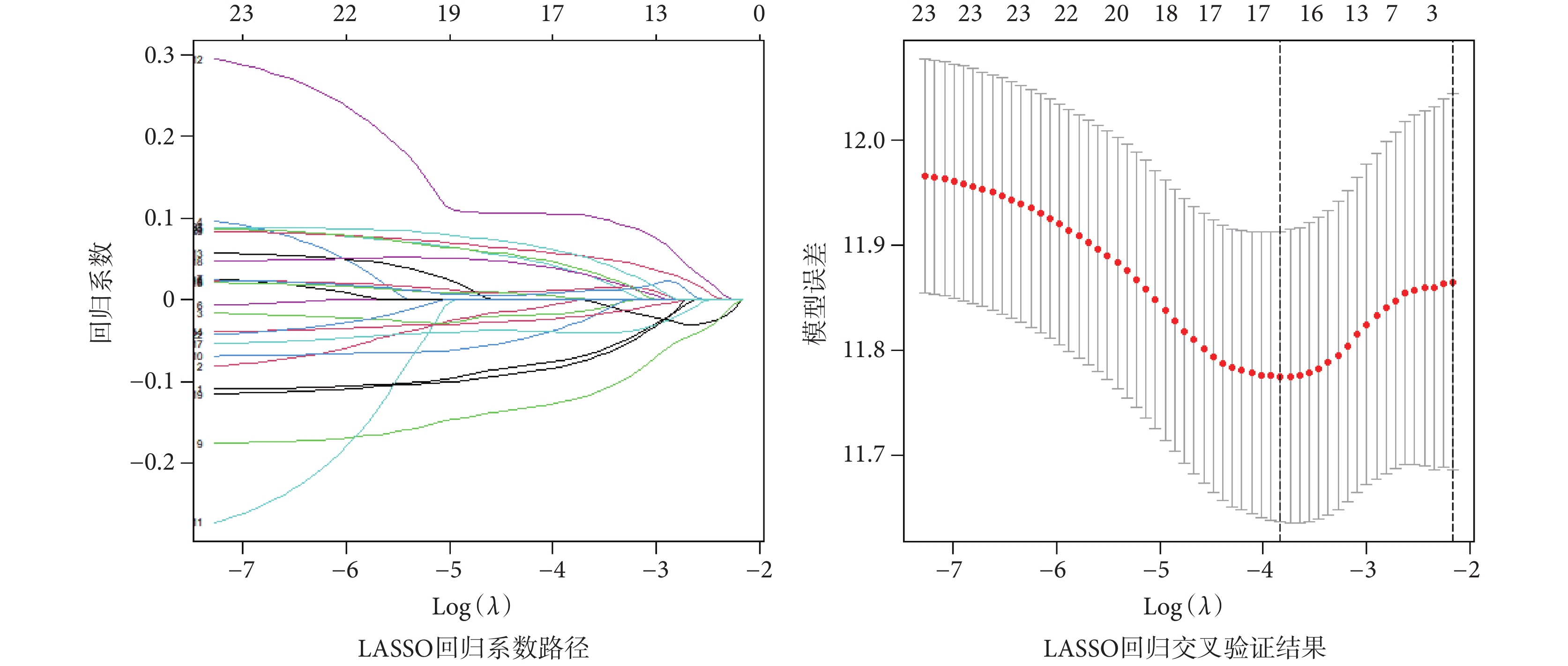
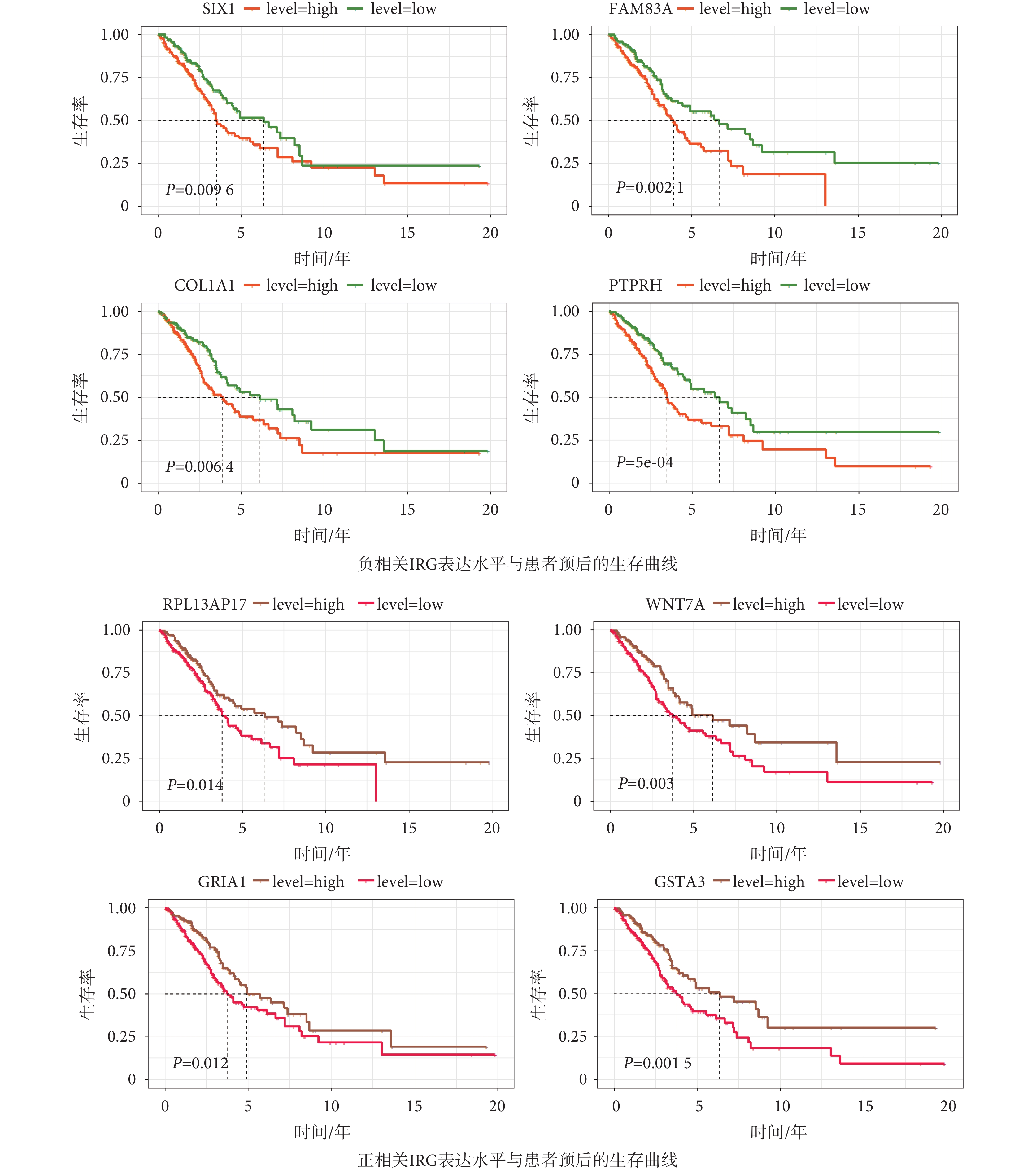
基于glmnet和survival包使用LASSO算法并進行十折交叉驗證進一步篩選用于建模的IRG(如圖2所示),最終得到可用于建模的IRG共17個。通過多因素CoxPH分析(如表2所示)發現,ADRB2、AGER、RPL13AP17、WNT7A、GRIA1、GSTA3與LUAD患者的生存呈正相關,TFAP2A、SIX1、KIF14、FAM83A、S100P、RHOV、AC025580.1、PCP4、PTPRH、COL1A1、TNFSF11與LUAD患者的生存呈負相關,且P值均小于0.05,表明這些IRG可作為LUAD患者的獨立預后因子。根據LUAD患者的生存期將患者劃分為高免疫風險組和低免疫風險組,進一步分析IRG與LUAD患者生存期的相關性,并使用survminer包繪制IRG表達水平與患者生存期的Kaplan Meier生存曲線。圖3展現了本文新發現的8個IRG與LUAD患者生存期的關系,通過生存曲線可看出篩選得到的IRG對LUAD患者的生存率有著顯著的分層效果。
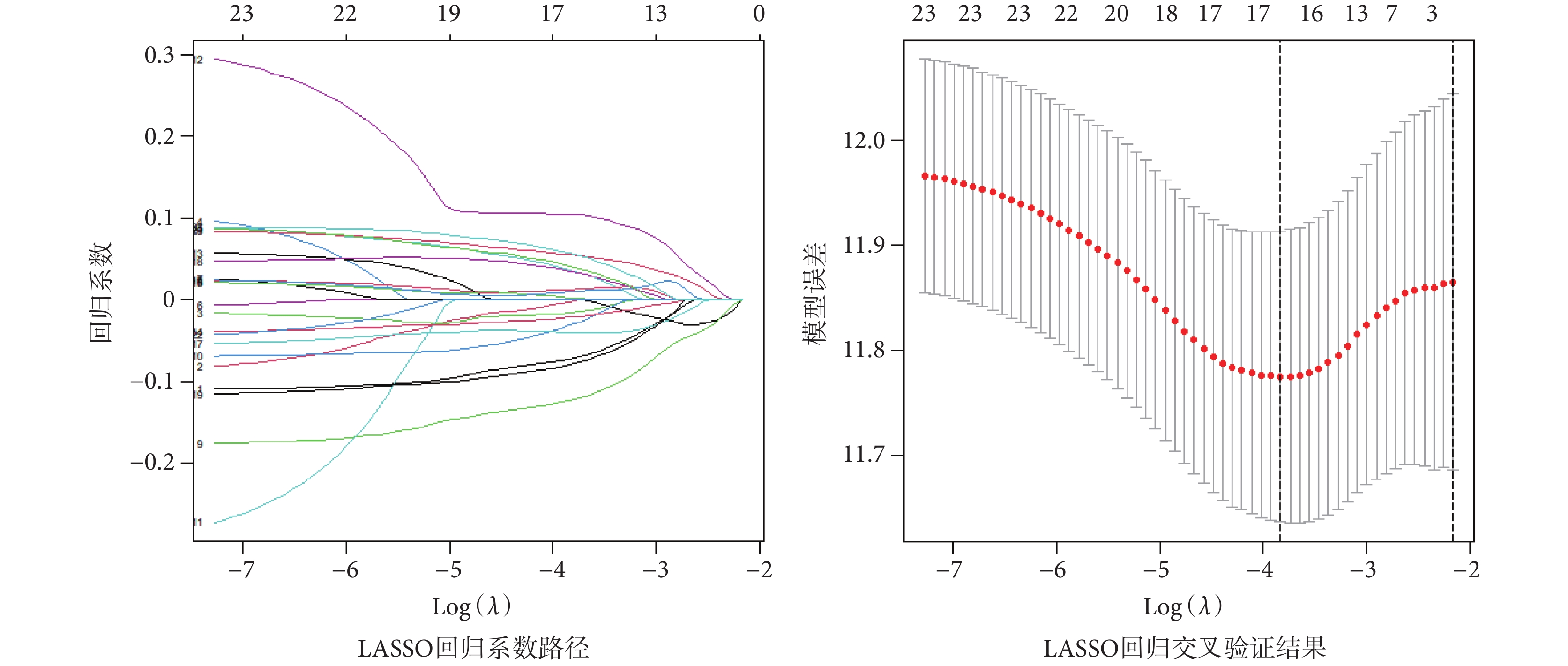 圖2
LASSO回歸分析的模型回歸系數圖與誤差圖
Figure2.
Model regression coefficient diagram and error diagram of LASSO regression analysis
圖2
LASSO回歸分析的模型回歸系數圖與誤差圖
Figure2.
Model regression coefficient diagram and error diagram of LASSO regression analysis
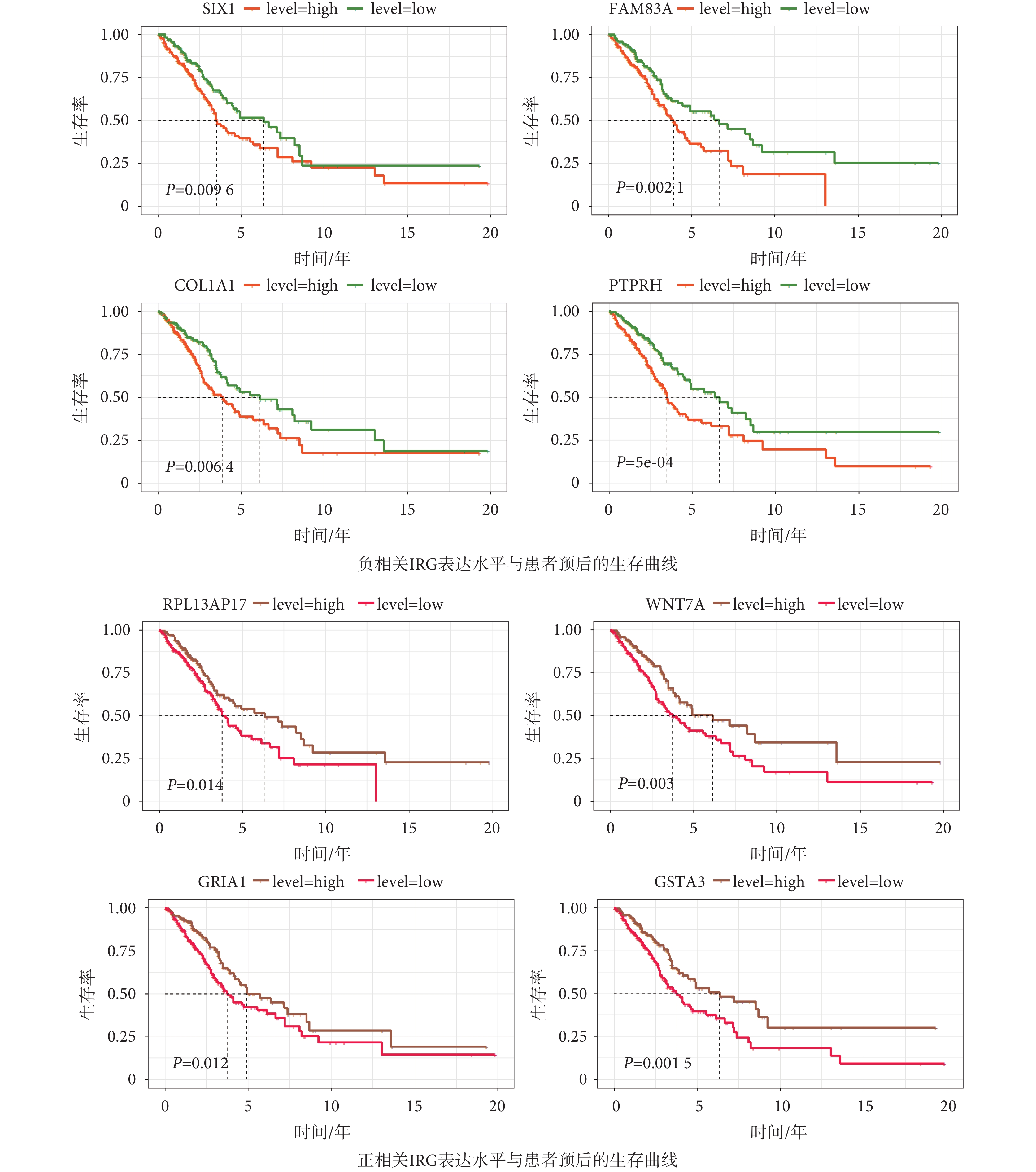 圖3
關鍵基因表達與患者預后的生存曲線
Figure3.
Survival curve of key gene expression and prognosis of patients
圖3
關鍵基因表達與患者預后的生存曲線
Figure3.
Survival curve of key gene expression and prognosis of patients
1.2.4 肺腺癌患者分層
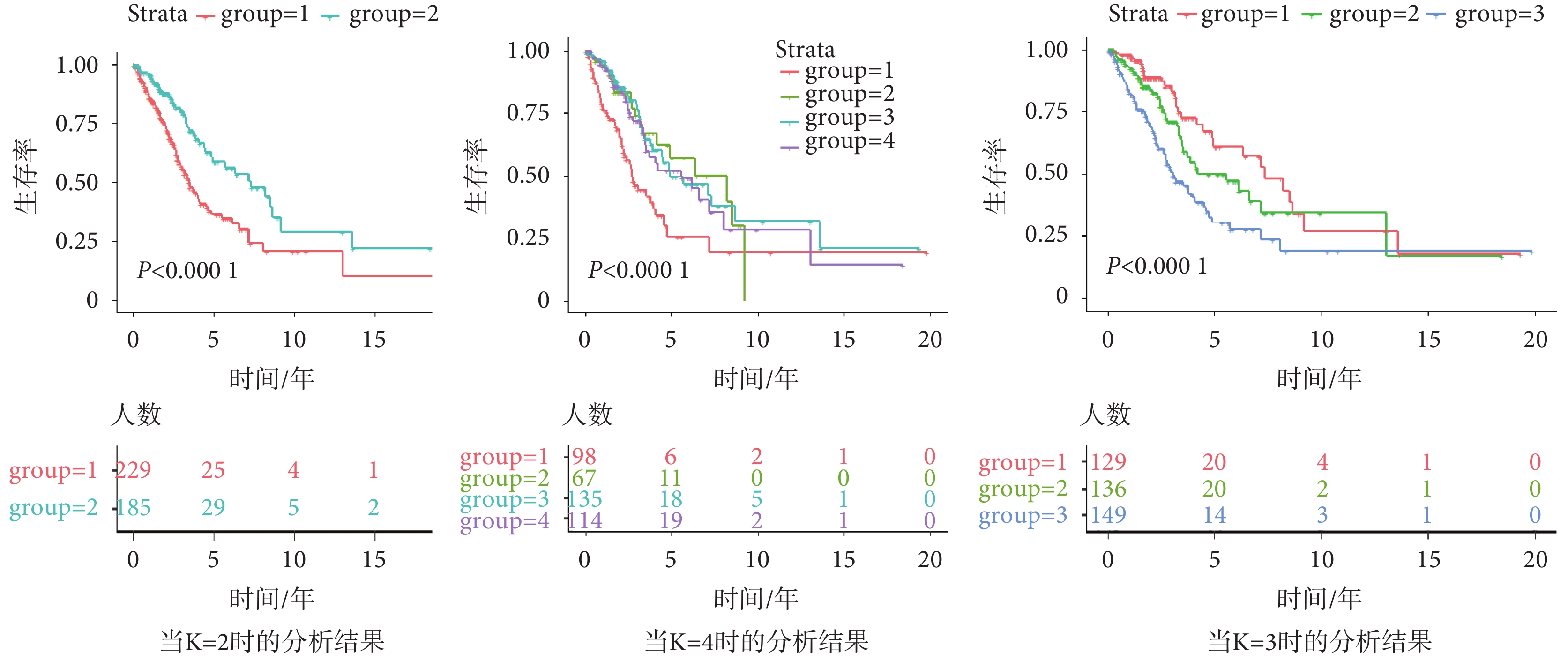
K-means聚類算法采用與生存相關的IRG對患者進行分層,分層的患者將被后續用于訓練預測模型[26-27]。在這項研究中,對K = 2~4時進行實驗(如圖4所示)。Kaplan Meier曲線顯示,當K = 2時,具有不同生存結局的患者被成功分離。但值得注意的是,當K = 3時,該算法成功地將患者分為三組,根據生存相關IRG特征,具有三種明顯不同的生存結果:5年生存率高于65%(第一組)、低于65%但高于30%(第二組)和低于30%(第三組))。
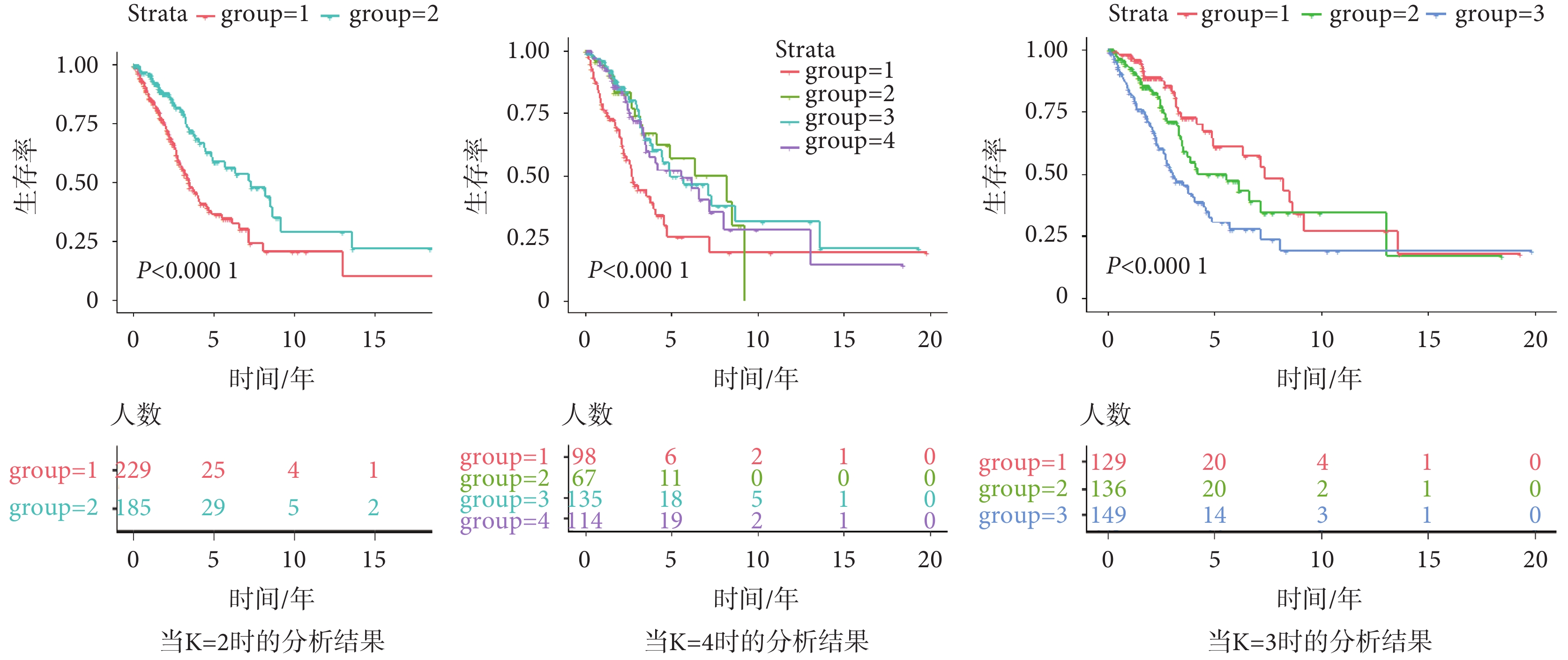 圖4
K-means回聚類分層LUAD患者的Kaplan Meier分析
Figure4.
Kaplan Meier analysis of LUAD patients stratified by K-means back clustering
圖4
K-means回聚類分層LUAD患者的Kaplan Meier分析
Figure4.
Kaplan Meier analysis of LUAD patients stratified by K-means back clustering
1.2.5 LightGBM預測模型
LightGBM是實現GBDT算法的框架,主要有以下幾個特點:Histogram算法、基于梯度的單邊采樣(gradient-based one-side sampling,GOSS)算法、特征綁定(exclusive feature bundling,EFB)算法、Leaf-wise的決策樹生長策略、高效并行及Cache命中率優化等。
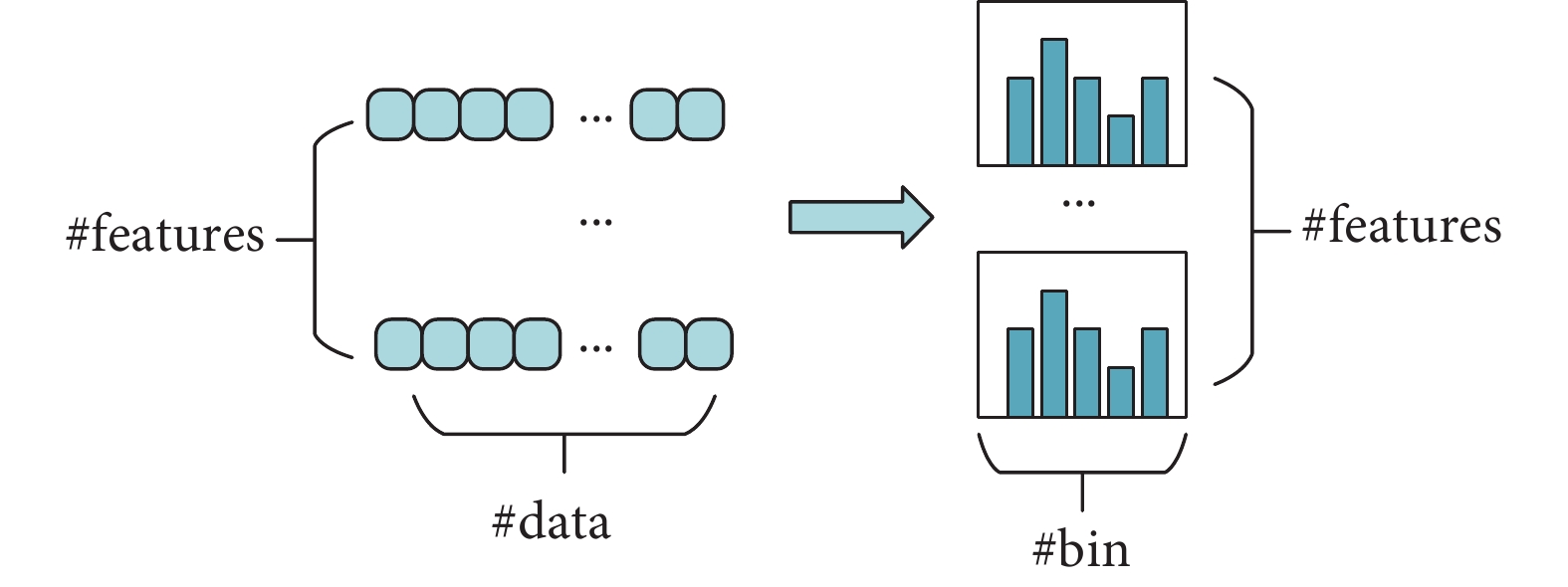
Histogram算法是對特征值進行裝箱處理,把連續的浮點型特征值離散化成k個整數,形成多個箱體(bin),同時構造一個寬為k的直方圖(如圖5所示)。在遍歷LUAD患者的IRG表達譜數據時先將浮點型的IRG表達值離散化,再把離散化后的值映射到相應的箱體中,當遍歷完一次數據后,對每一個箱體中的數據進行統計并繪制直方圖,然后根據直方圖的離散值,遍歷尋找最優分割點。這樣在訓練模型時就可直接使用離散化后的統計值,而不是原始的浮點型數據。這樣做的好處是減少特征的取值空間,從而提高模型的訓練效率和性能。此外,離散化后的特征也更便于進行特征的交叉和組合,以進一步提升模型的性能。值得注意的是,Histogram使用箱體代替原始數據,相當于增加了正則化,這意味著有更多的細節特征會被丟棄,相似的數據被分到同一個箱體中,所以箱體的數量決定了正則化程度,箱體越少懲罰越嚴重,過擬合的風險越低。
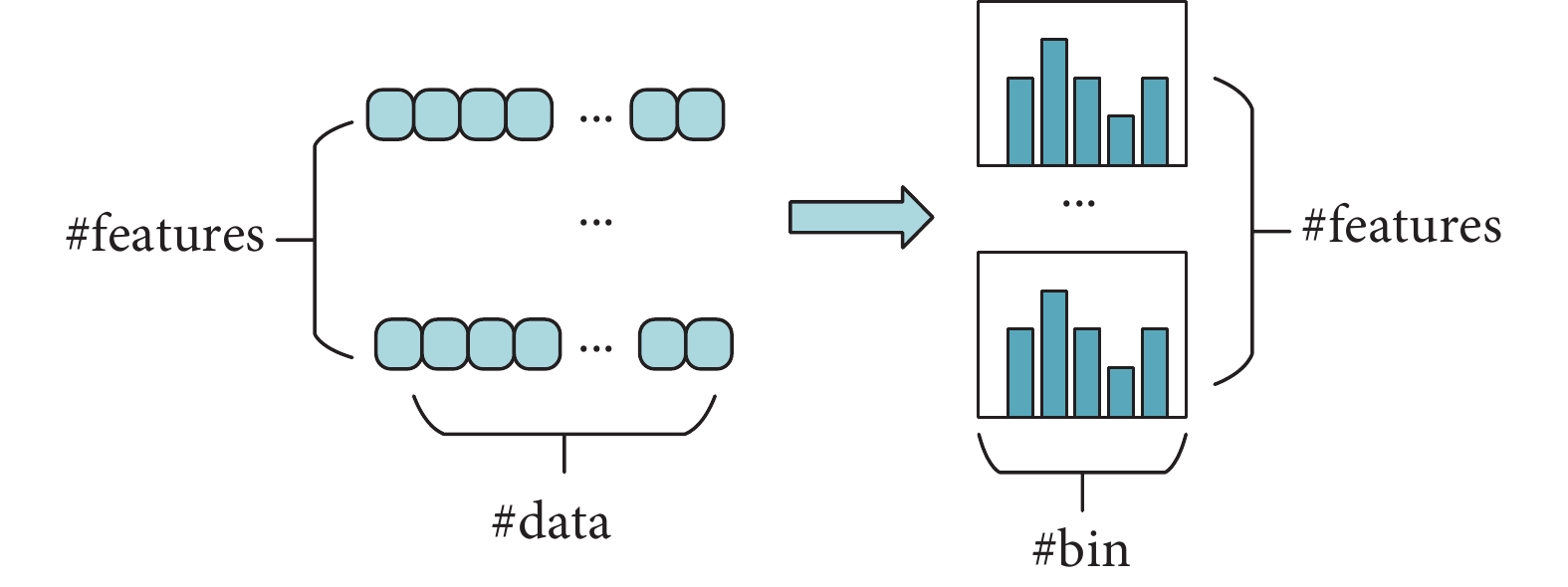 圖5
Histogram算法裝箱處理過程
Figure5.
Histogram algorithm boxing process
圖5
Histogram算法裝箱處理過程
Figure5.
Histogram algorithm boxing process
GOSS算法通過對樣本采樣的方法來減少計算目標函數增益時的復雜度。在本實驗中,首先計算每個樣本的梯度并進行排序,然后選取梯度最大的a*100%個樣本作為集合A(實驗中a = 0.2),然后在剩下的數據中隨機采樣b*100%個樣本作為集合B(實驗中b = 0.3),之后計算信息增益時只在A∪B上運算,但為了保持數據分布不變,通常乘上系數(1 – a)/b來補償小梯度樣本。最后根據計算得到的信息增益對特征進行排序,以了解哪些IRG對預測結果有更大的影響。
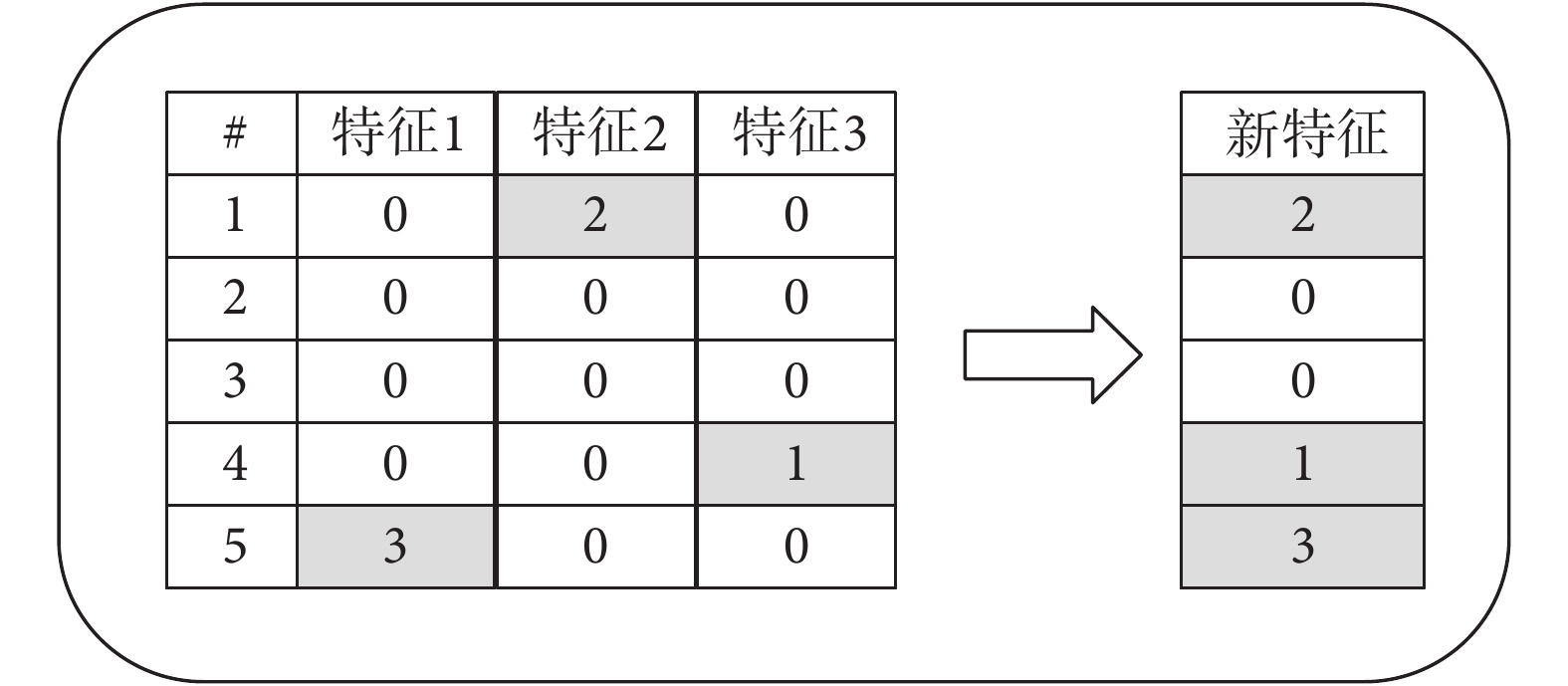
EFB算法是處理現實生活中常見的高維數據、稀疏特征空間的方法(如圖6所示)。設特征1、特征2及特征3互為互斥的稀疏特征,通過EFB算法將三個特征捆綁為一個稠密的新特征,然后用該新特征替代原來的三個特征,從而實現不損失信息的情況下減少特征維度,提升梯度增強算法的速度。如本文中在使用LightGBM模型與XGBoost模型進行聯合分析篩選與LUAD預后相關的IRG時,就是將IRG表達譜數據隨機分組,然后對每一組數據進行分析,探尋IRG之間的相關性。如果當一個或幾個IRG表達值上升或下降時,其余的IRG表達值也隨之上升或下降,那么說明這些IRG之間具有相關性,此時選擇最具代表性的幾個IRG作為與LUAD預后相關的主要IRG特征進行保留。經過反復的實驗驗證后,對實驗結果取交集便初步篩選到了與LUAD預后相關的IRG。
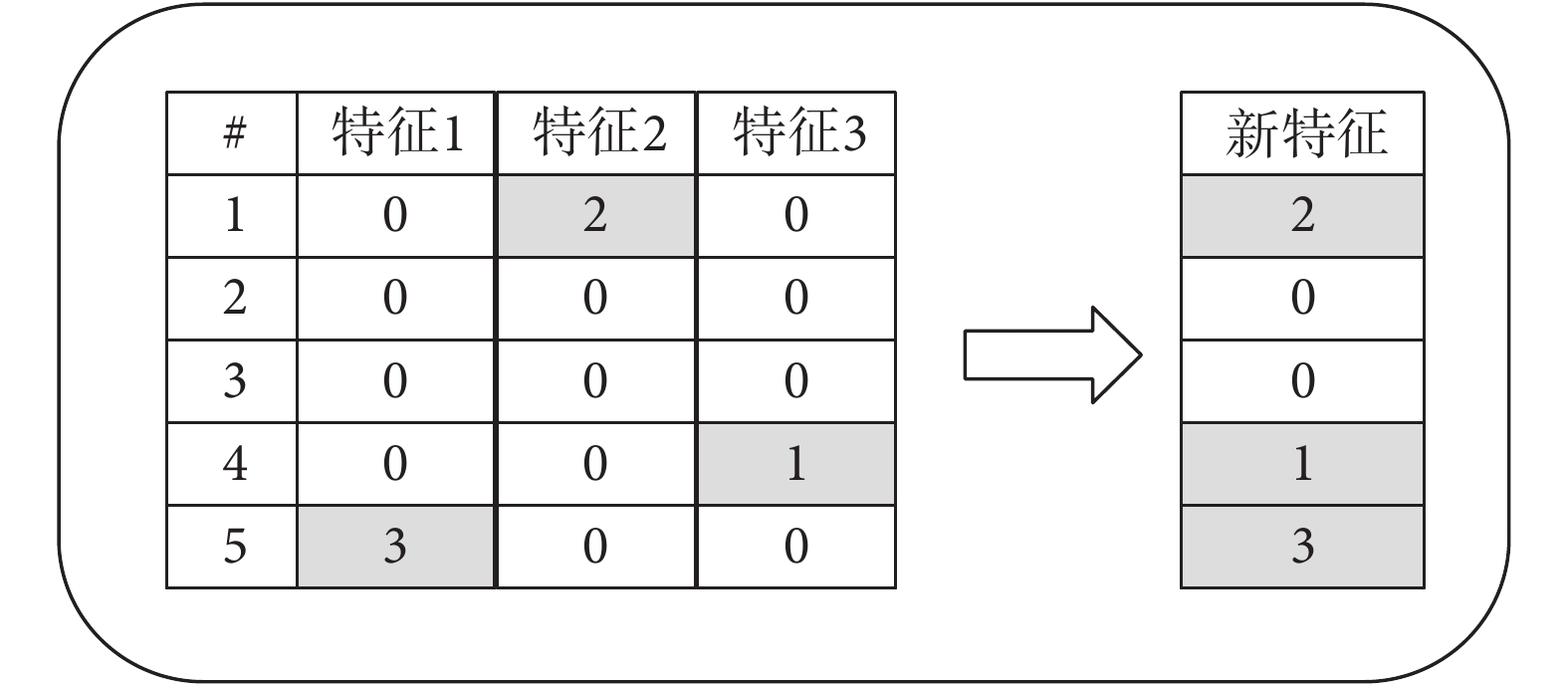 圖6
EFB算法特征處理過程
Figure6.
Feature processing process of EFB algorithm
圖6
EFB算法特征處理過程
Figure6.
Feature processing process of EFB algorithm
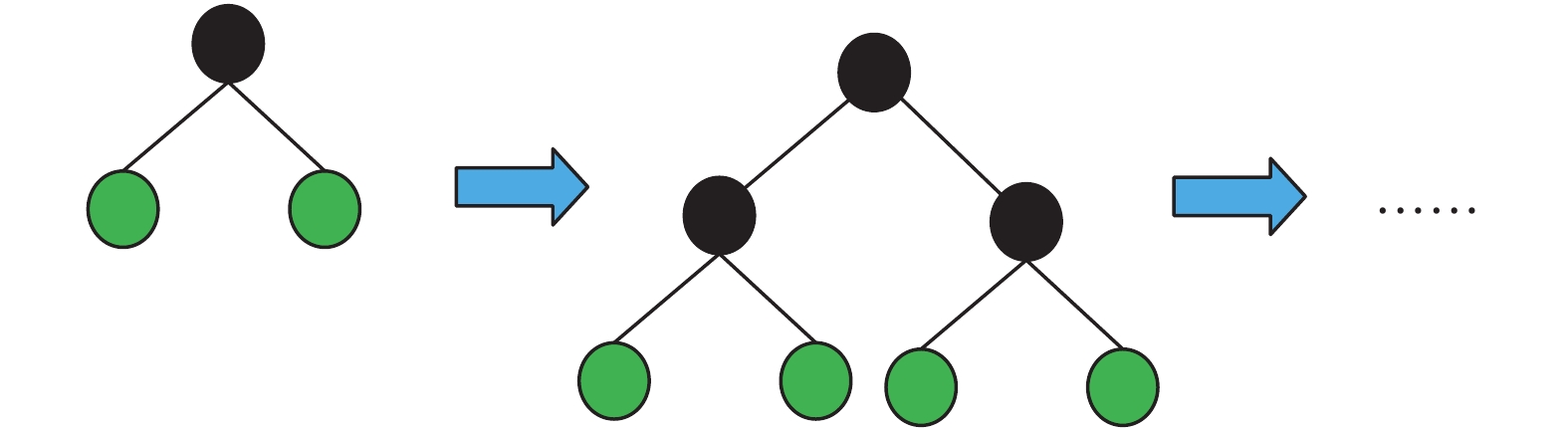
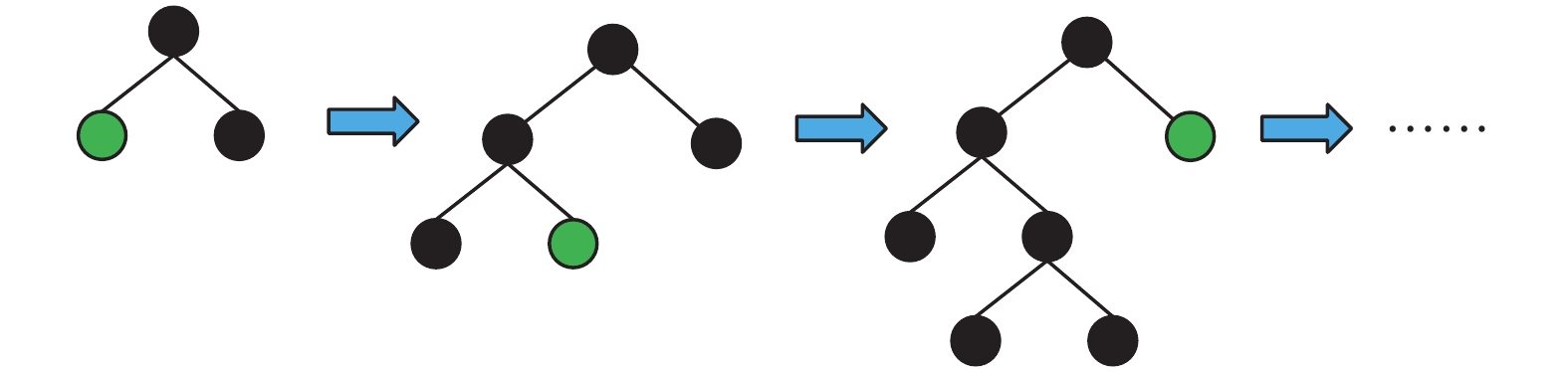
大部分決策樹的學習算法采用Level-wise策略生長樹,不加區分地一次分裂同一層的所有葉子,但很多葉子的分裂增益較低,所以該策略會產生許多不必要的分裂,消耗了更多的運行內存(如圖7所示)。LightGBM算法對它進行了改進,使用Leaf-wise策略生長樹,即每次只對分裂增益最大的葉子進行分裂,其他葉子不變。Leaf-wise策略可降低誤差,得到更好的精度,并能加快算法的學習速度(如圖8所示)。
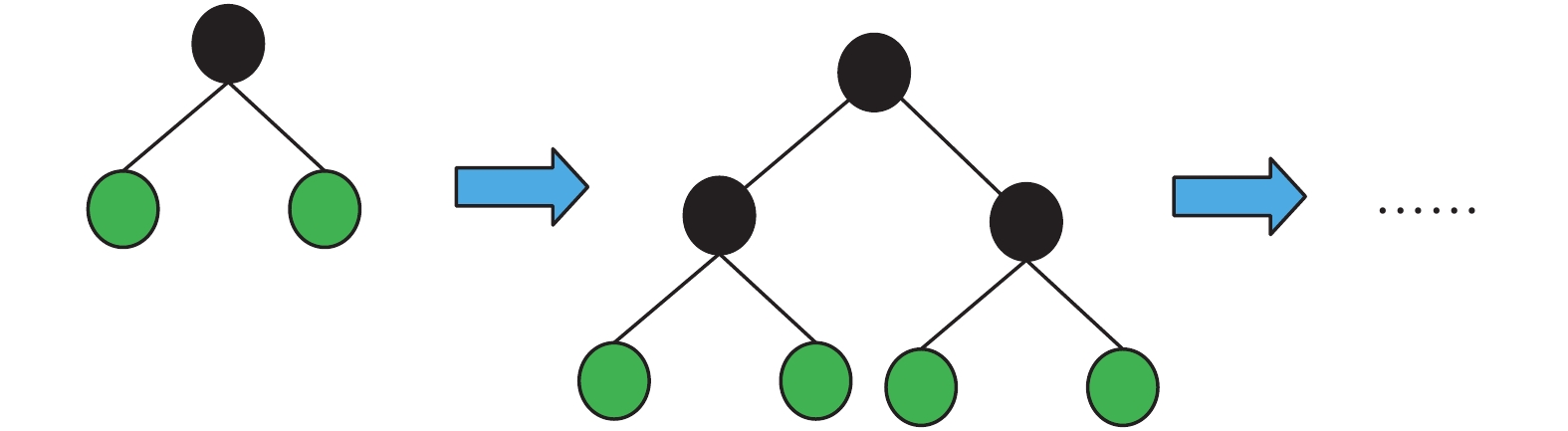 圖7
Level-wise策略生長樹
Figure7.
Level-wise strategy growth tree
圖7
Level-wise策略生長樹
Figure7.
Level-wise strategy growth tree
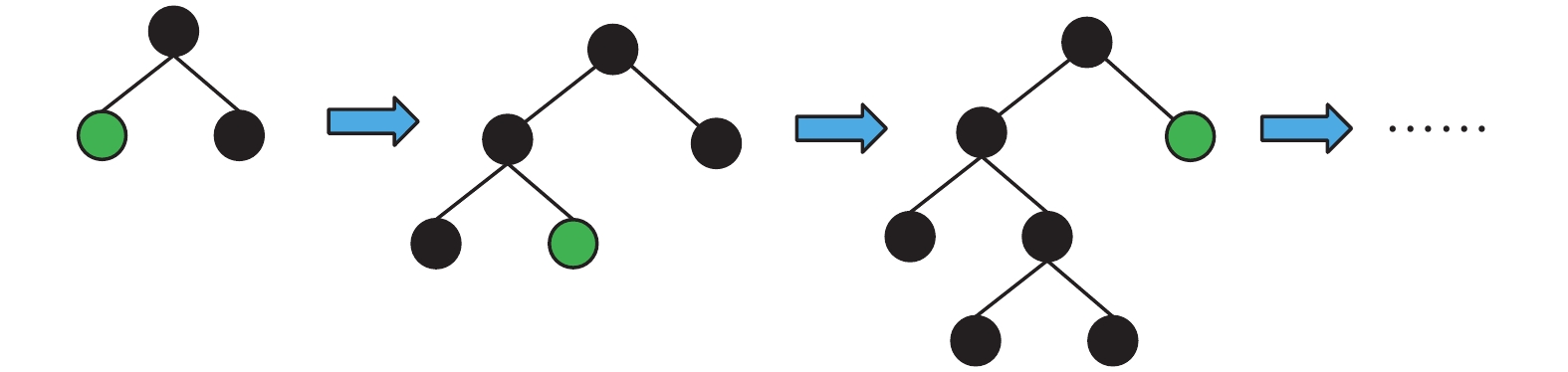 圖8
Leaf-wise策略生長樹
Figure8.
Leaf-wise strategy growth tree
圖8
Leaf-wise策略生長樹
Figure8.
Leaf-wise strategy growth tree
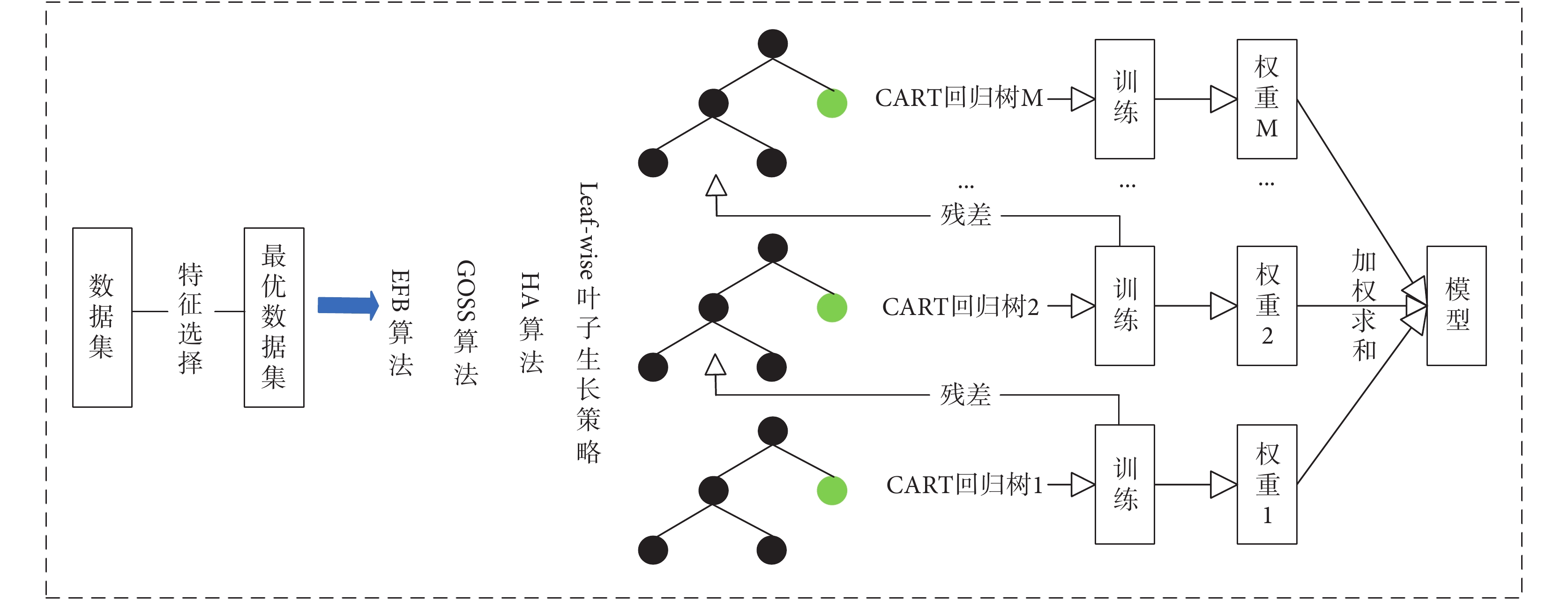
總的來說,LightGBM是一個性能高度優化的GBDT算法,將LightGBM的優化用公式表達為LightGBM = XGBoost + Histogram + GOSS + EFB(如圖9所示)。其中,Histogram是直方圖算法,作用是減少候選分類點;GOSS是基于梯度的單邊采樣算法,作用是減少樣本數量;EFB是互斥特征捆綁算法,作用是減少特征數量。LightGBM支持高效并行訓練,且具有更快的訓練速度、更低的內存消耗、更好的準確率以及支持分布式快速處理海量數據等優點。
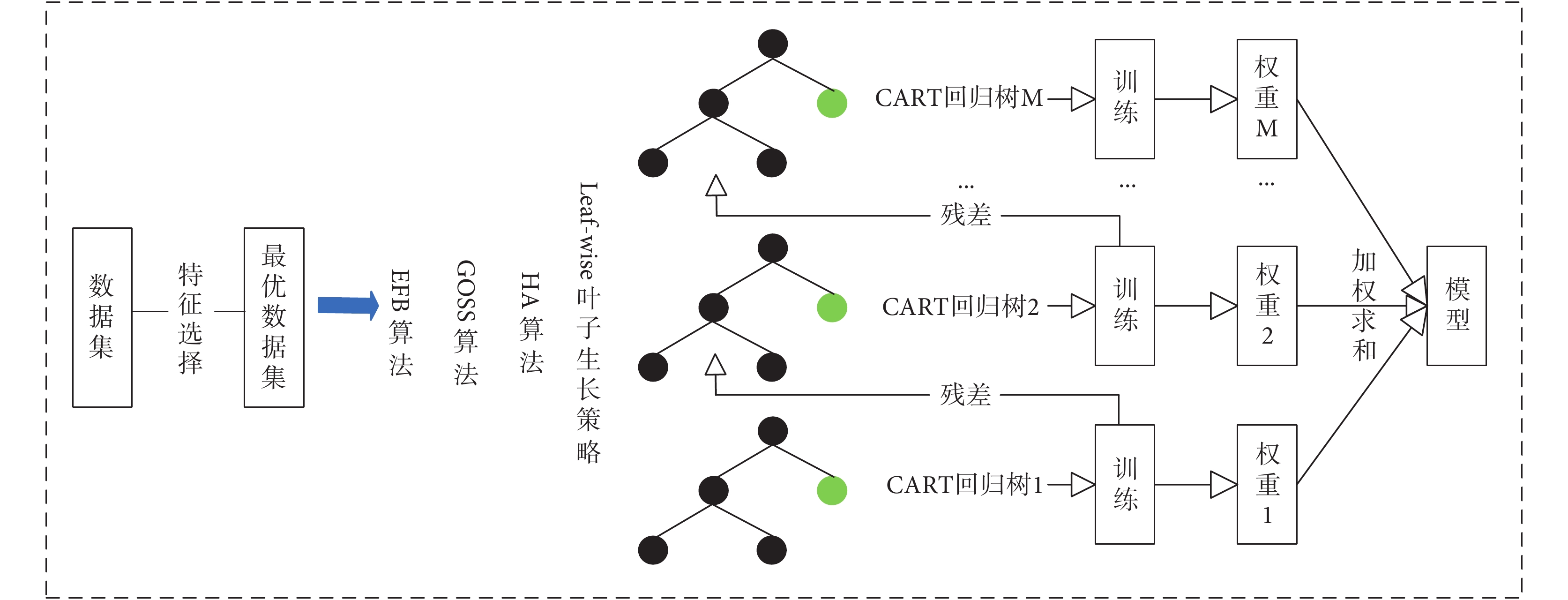 圖9
LightGBM模型結構
Figure9.
LightGBM model structure
圖9
LightGBM模型結構
Figure9.
LightGBM model structure
將患者的K-means聚類結果和IRG表達譜數據納入LightGBM模型,用于預后模型的訓練和預測。在該項研究中,使用7/3拆分來生成訓練數據集和測試數據集,并選擇十折交叉驗證(cross-validation,CV)。
2 結果
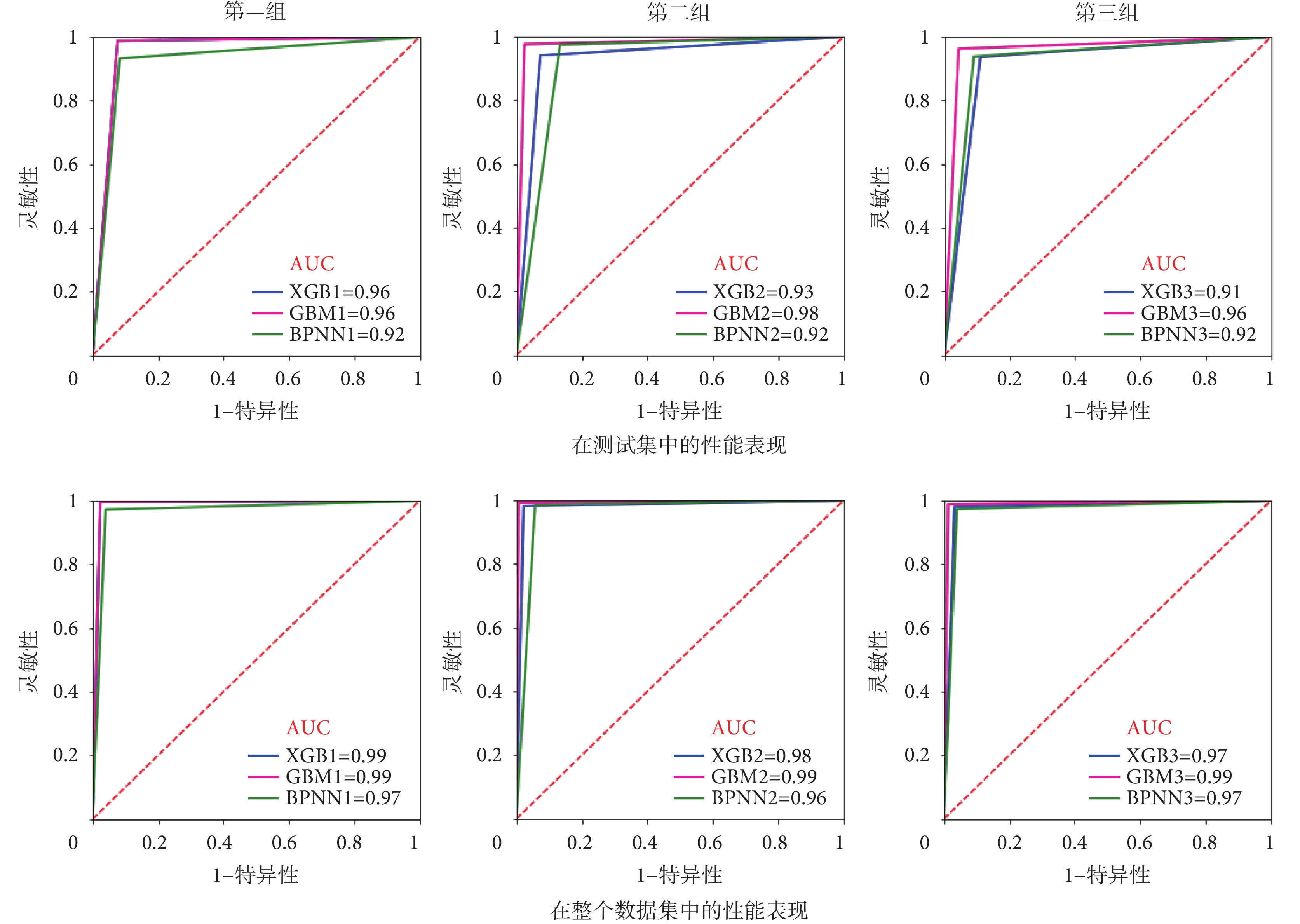
本文使用XGBoost集成學習模型和BPNN深度學習模型作為基線模型。模型訓練完成后,使用受試者操作特征(receiver operating characteristic,ROC)曲線下面積(area under curve,AUC)來評估模型的效果(如圖10所示)。在測試集中,LightGBM模型的AUC值分別為0.96(第一組)、0.98(第二組)、0.96(第三組);在整個數據集中,LightGBM模型的AUC值分別為0.99(第一組)、0.99(第二組)、0.99(第三組)。在測試集中,對于第一組患者的生存率預測,XGBoost與LightGBM達到了相同的準確率(0.96),BPNN相對較差,僅為0.92;對于第二組和第三組患者的生存率預測,LightGBM模型預測效果優于XGBoost和BPNN模型。在整個數據集中,也只有在對第一組患者進行生存預測時,XGBoost模型能與LightGBM達到相同的準確率,而對于其他組患者的生存預測,LightGBM模型的效果優于XGBoost和BPNN模型。此外,通過LightGBM模型預測的患者表現出與通過K-means聚類的患者相似的生存結果,這也證實了LightGBM預測模型的準確性。本文提出的基于LightGBM的集成學習方法可將LUAD患者分為高生存率(5年生存率 ≥ 65%)、中等生存率(5年生存率 < 65%且 ≥ 30%)和低生存率(5年生存率 < 30%)三個組。
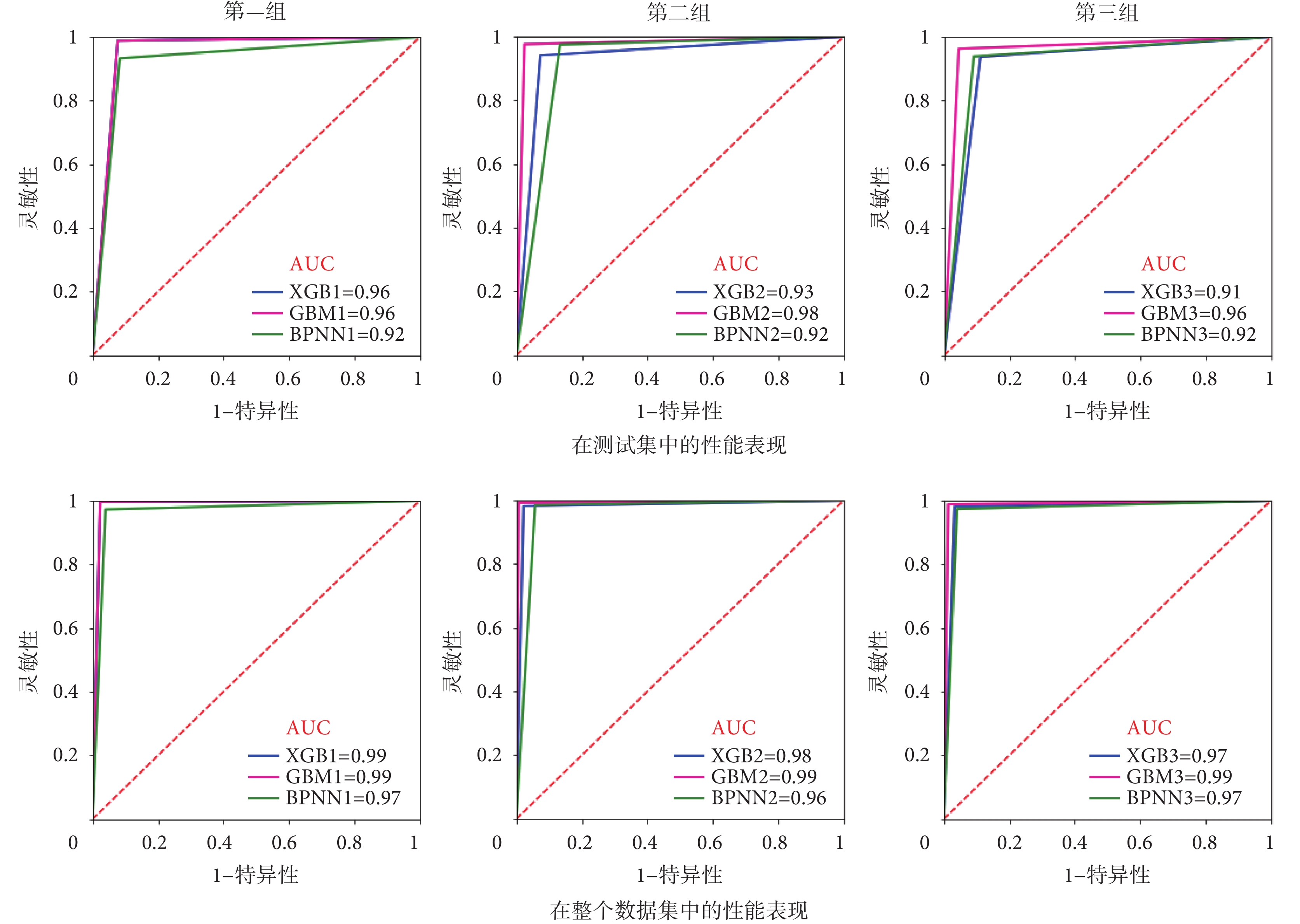 圖10
模型性能的評估
Figure10.
Evaluation of model performance
圖10
模型性能的評估
Figure10.
Evaluation of model performance
3 討論
腫瘤異質性包括腫瘤間異質性(不同腫瘤細胞之間的基因與表現型不同)和腫瘤內異質性(相同腫瘤細胞內的基因與表現型不同),其中腫瘤內異質性又有空間異質性(相同腫瘤不同區域)和時間異質性(原發腫瘤和繼發腫瘤)之分[28]。因此,理想情況下,缺失值應替換為相同亞型腫瘤樣本中得出的平均值。由于數據來源是RNA-seq轉錄組測序,理論上,所有的基因都會被檢錄,絕大部分沒有被檢錄到的基因是因為它們的表達水平過低。所以,一般對轉錄組數據補缺失值的方法采用補0法,而我們采用的KNN方法是一種基于相似性的方法,它考慮了樣本之間的相關性,能更好地保留數據的整體結構。
準確預測癌癥患者的生存率對于治療決策十分重要。近年來,已有研究結果表明,IRG在LUAD的發生發展過程中發揮著重要的調控作用[29-31]。基于分子特征預測模型的開發為提高癌癥生存預測的準確性帶來了希望。但是,目前大多數基于分子的生存預測模型都是將患者分為兩組,其生存結果不同[32]。例如黃正品等[10]提出的LASSO-Cox模型和劉鳳燕等[33]提出的多因素Cox模型,將LUAD患者分為高低風險組,五年生存率預測(AUC值)分別為0.74和0.72。然而,這一結果對于治療決策來說不夠準確。在本研究中,使用LightGBM模型將LUAD患者分為三組,五年生存率預測AUC為0.96。
本研究從TCGA下載肺腺癌的RNA-seq數據和臨床數據,通過生物信息學方法和集成學習方法進行分析和挖掘,得到了17個IRG,其中KIF14、RHOV、S100P、AC025580.1、PCP4、TFAP2A、TNFSF11、ADRB2、AGER等IRG在早期的研究中已被提出[34-36]。本研究使用LightGBM模型新發現COL1A1、SIX1、FAM83A、PTPRH、RPL13AP17、WNT7A、GRIA1、GSTA3等8個IRG。預后分析結果顯示,這些IRG均與LUAD的預后相關(如圖3所示)。此外,有研究報道了COL1A1在NSCLC中表達高于正常組織[37],本研究的分析結果表明COL1A1與LUAD患者預后生存期呈負相關,更加驗證了COL1A1對LUAD的發生發展具有重要意義。
綜上所述,本研究分析新發現的8個IRG在LUAD組織與正常組織中的表達存在顯著差異,對LUAD預后評估有一定的參考價值,可作為LUAD的生物標志物進一步研究。因此,本研究的發現將有助于從分子機制了解LUAD的發生發展,并為LUAD的早期診斷、靶點治療和預后評估提供新的潛在生物學標志物。
4 結論
在這項研究中,我們基于TCGA數據庫和LightGBM集成學習方法構建LUAD患者的生存率預測模型,首次將LightGBM模型引入LUAD預后分析,成功克服了以往線性模型無法準確描述數據之間復雜非線性關系的局限,也克服了深度學習模型難以解釋的不足。通過利用非線性函數進行數據擬合,能夠更加精準地揭示數據之間的聯系,從而得到更精確的預測結果。該模型能夠將LUAD患者分為三組:5年生存率高于65%(第1組)、低于65%但高于30%(第2組)和低于30%(第3組),并且能較準確地預測LUAD患者的五年生存率。本模型的應用不僅提高了預測的準確性,而且使得結果更具可解釋性和可理解性,為醫生和患者提供了更可靠的參考依據。
但該模型也還存在一定的局限。首先,本研究從公共數據庫TCGA中獲取LUAD的相關數據,仍需要在多中心的大規模患者隊列中進一步驗證該模型的性能。此外,由于是基于生物信息學分析的研究,所以一些基因的預后性能需要進行相關的實驗來進一步證實。同時,雖然本研究是基于轉錄組測序數據進行的,理論上該模型具有應用于其他腫瘤預后評估的潛力,然而其實際表現尚未得到充分驗證,仍需進一步的研究。綜上所述,未來應該致力于驗證該模型在更大規模患者隊列中的表現,并通過實驗驗證相關基因的功能和作用機制,以進一步提高該模型的可靠性和準確性。另一方面還可以探索該模型在其他腫瘤預后評估中的應用潛力,為臨床實踐提供更多有效的工具。
重要聲明
利益沖突聲明:本文全體作者均聲明不存在利益沖突。
作者貢獻聲明:孟祥福主要負責總體解決方案設計、實驗指導、論文撰寫、論文審閱及修訂;田友發主要負責實驗方案設計、程序編寫、論文撰寫;張霄雁主要負責算法咨詢與建議。
0 引言
肺癌是世界上對人類生命健康威脅最大的惡性腫瘤之一,其發病率和死亡率已分別占到癌癥總發病率和總死亡率的11.6%和18.4%[1]。據2020年世界衛生組織國際癌癥研究機構發布的最新數據顯示,中國2020年新發肺癌83萬例,死亡人數71萬,成為世界第一肺癌大國[2-3]。此外,非小細胞肺癌(non-small cell lung cancer,NSCLC)占所有肺癌的85%以上,其中肺腺癌(lung adenocarcinoma,LUAD)的發生率最高,約占所有NSCLC的50%[4-5]。LUAD早期癥狀不明顯,確診時常處于晚期,從而錯過最佳手術時間,導致LUAD患者五年生存率一直較低,僅為15%左右;但也有數據顯示,在早期被診斷出LUAD的患者通過積極治療,其五年生存率接近100%[6-7]。
目前,LUAD臨床預后的判斷方法主要是美國癌癥聯合會(American Joint Committee on Cancer,AJCC)提出的TNM(tumor node metastasis)分期系統[8]。TNM分期系統使用三個值來描述癌癥的進展:T表示腫瘤的大小,N表示轉移至淋巴結,M表示轉移至遠處器官,較高的數字代表癌癥的更晚期。但該分期系統只考慮了腫瘤的解剖因素,而沒有考慮非解剖因素,所以不能準確地預測LUAD的預后[9]。
免疫相關基因(immune related gene,IRG)在LUAD的發生發展過程中發揮著重要的調控作用,探索異常表達的IRG并構建預后生物標志物,一方面有利于對LUAD發病機制的理解,另一方面對制定個體化診療方案以及發現新的治療靶標等具有重要意義[10]。在傳統的生存分析研究中,Cox比例風險模型(Cox proportional hazard model,CoxPH)是最常用的半參數回歸模型,其基本思想是根據患者性別、吸煙年限和身體質量指數(body mass index,BMI)等結構化特征進行生存分析。雖然回歸模型的每個變量易于解釋,但表達協變量之間的非線性關系是有限的,且無法學習圖像數據等非結構化數據。因此,集成學習和深度學習方法常用來克服線性模型的缺點[11-13]。在深度學習方面,Anika等[14]構建了基于多模態神經網絡的模型,利用臨床數據和基因表達譜數據預測20種不同癌癥類型患者的生存率。Zhu等[15]提出一種使用病理圖像進行生存分析的深度卷積神經網絡學習圖像深層次的信息,從而提高生存預測性能。在集成學習方面,Thedinga等[16]引入XGBoost(eXtreme Gradient Boosting)模型,對來自25種不同癌癥類型的8 024名患者的轉錄組數據進行患者生存率預測。
轉錄組學技術(微陣列、RNA測序)提供了關于患者的大量分子特征信息,對這些分子特征數據進行分析,進而預測癌癥患者生存率是生物醫學研究的一個重要內容[17]。在LUAD的發生發展過程中,癌細胞的變化會破壞基因的表達,從而留下有價值的信息,這些信息是多種基因表達的非線性組合。
本研究的目的是構建一種新型的基于分子特征的集成學習LUAD患者生存率預測模型。選擇IRG作為特征,分別使用Limma包、CoxPH、最小絕對收縮和選擇算子(least absolute selection and shrinkage operator,LASSO)、K-means聚類、XGBoost、LightGBM(Light Gradient Boosting Machine)和反向傳播神經網絡(back propagation neural network,BPNN)等算法進行生存相關IRG特征的識別、IRG特征的目標分層和預測模型的構建[18-25],以期提高LUAD預后預測模型的準確性,進而輔助醫生為患者提供個體化治療方案。
1 材料和方法
1.1 材料
本文使用的數據來源于癌癥基因組圖譜(The Cancer Genome Atlas,TCGA)。該數據庫是由美國國家癌癥研究所和美國國家人類基因組研究所于2006年聯合啟動建立的項目,旨在應用高通量的基因組分析技術,幫助人們對癌癥有更好的認知,從而提高對癌癥的預防、診斷和治療能力[2]。TCGA收錄了各種人類癌癥的組學數據,包括基因表達數據、拷貝數變異和DNA甲基化等。該數據庫也是癌癥研究者很重要的數據來源,目前已收錄了11 000多例患者和33種癌癥的數據。
本文采用Python整合從TCGA數據庫(https://portal.gdc.cancer.gov)下載的RNA-seq數據和臨床數據。從RNA-seq數據中獲取gene_id、gene_name和unstranded列數據來構成IRG表達數據,獲得的IRG表達數據包含510例樣本(45例正常樣本、450例肺腺癌樣本和15例轉移組樣本)和60 660個轉錄組測序結果。臨床數據是兩個Json文件,從中提取出樣本的生存時間、生存狀態、性別、AJCC分期、TNM分期((T、N、M)等數據。
1.2 方法
本文提出的方法分為3步:首先對IRG表達譜數據和臨床數據進行預處理,得到與LUAD診斷相關的IRG;然后利用K-means聚類算法對LUAD患者進行分層;最后構建基于LightGBM的肺腺癌預測模型并根據第一步得到的IRG數據和第二步的患者分層數據,對患者的生存率進行預測。源代碼在GitHub中提供(https://github.com/tianyoufa/tianyoufaCode)。
1.2.1 數據預處理
從數據中移除15例轉移組樣本,采用K最近鄰(K-nearest neighbors,KNN)算法填充缺失值,與從IMMPORT數據庫下載的免疫基因取交集,提取出與肺腺癌相關的差異表達免疫基因共2 287個。剔除在癌癥組與正常組中IRG表達量均值之比在區間[0.8, 1.2]內的IRG數據,因為該區間內的IRG在患病前與患病后無明顯變化,說明該IRG對是否患LUAD沒有影響。將臨床數據與IRG表達譜數據整合,更新患者的診斷數據,移除重復出現的患者數據,使用最新的檢測結果代替。經過預處理后的414例LUAD患者臨床數據如表1所示。
1.2.2 篩選差異表達的IRG
基于R語言及Limma包,使用log2轉化后的IRG表達譜數據對45例正常組織和414例LUAD組織進行基因差異表達分析,差異表達基因的選擇條件為fdr < 0.05且|logFC| > 2。篩選出差異基因共647個,包括上調基因348個,下調基因299個。
1.2.3 與預后相關的IRG篩選方法
由于樣本量較少而基因較多,導致在高維空間中數據存在一定冗余信息和噪聲信息,這些信息會在建模過程中引入誤差,而對數據降維可降低模型的復雜度并提高模型的精度。因此,在得到差異表達分析結果后,先對差異表達基因以P < 0.1為閾值進行單因素CoxPH回歸分析,對數據進行降維,再以P < 0.05為閾值進行多因素CoxPH回歸分析,并對實驗結果取交集得到與預后相關的79個IRG。將CoxPH回歸分析得到的IRG輸入XGBoost與LightGBM模型進行實驗分析,對IRG的分析結果進行打分,再對XGBoost的兩組實驗結果取交集,對LightGBM的兩組實驗結果取交集,最后將XGBoost與LightGBM的實驗結果取并集,最終得到23個與預后顯著相關的IRG(如圖1所示)。
 圖1
多因素Cox回歸分析與XGBoost和LightGBM聯合分析結果的韋恩圖
Figure1.
Venn diagram of multivariate Cox regression analysis and joint analysis of XGBoost and LightGBM
圖1
多因素Cox回歸分析與XGBoost和LightGBM聯合分析結果的韋恩圖
Figure1.
Venn diagram of multivariate Cox regression analysis and joint analysis of XGBoost and LightGBM
基于glmnet和survival包使用LASSO算法并進行十折交叉驗證進一步篩選用于建模的IRG(如圖2所示),最終得到可用于建模的IRG共17個。通過多因素CoxPH分析(如表2所示)發現,ADRB2、AGER、RPL13AP17、WNT7A、GRIA1、GSTA3與LUAD患者的生存呈正相關,TFAP2A、SIX1、KIF14、FAM83A、S100P、RHOV、AC025580.1、PCP4、PTPRH、COL1A1、TNFSF11與LUAD患者的生存呈負相關,且P值均小于0.05,表明這些IRG可作為LUAD患者的獨立預后因子。根據LUAD患者的生存期將患者劃分為高免疫風險組和低免疫風險組,進一步分析IRG與LUAD患者生存期的相關性,并使用survminer包繪制IRG表達水平與患者生存期的Kaplan Meier生存曲線。圖3展現了本文新發現的8個IRG與LUAD患者生存期的關系,通過生存曲線可看出篩選得到的IRG對LUAD患者的生存率有著顯著的分層效果。
 圖2
LASSO回歸分析的模型回歸系數圖與誤差圖
Figure2.
Model regression coefficient diagram and error diagram of LASSO regression analysis
圖2
LASSO回歸分析的模型回歸系數圖與誤差圖
Figure2.
Model regression coefficient diagram and error diagram of LASSO regression analysis
 圖3
關鍵基因表達與患者預后的生存曲線
Figure3.
Survival curve of key gene expression and prognosis of patients
圖3
關鍵基因表達與患者預后的生存曲線
Figure3.
Survival curve of key gene expression and prognosis of patients
1.2.4 肺腺癌患者分層
K-means聚類算法采用與生存相關的IRG對患者進行分層,分層的患者將被后續用于訓練預測模型[26-27]。在這項研究中,對K = 2~4時進行實驗(如圖4所示)。Kaplan Meier曲線顯示,當K = 2時,具有不同生存結局的患者被成功分離。但值得注意的是,當K = 3時,該算法成功地將患者分為三組,根據生存相關IRG特征,具有三種明顯不同的生存結果:5年生存率高于65%(第一組)、低于65%但高于30%(第二組)和低于30%(第三組))。
 圖4
K-means回聚類分層LUAD患者的Kaplan Meier分析
Figure4.
Kaplan Meier analysis of LUAD patients stratified by K-means back clustering
圖4
K-means回聚類分層LUAD患者的Kaplan Meier分析
Figure4.
Kaplan Meier analysis of LUAD patients stratified by K-means back clustering
1.2.5 LightGBM預測模型
LightGBM是實現GBDT算法的框架,主要有以下幾個特點:Histogram算法、基于梯度的單邊采樣(gradient-based one-side sampling,GOSS)算法、特征綁定(exclusive feature bundling,EFB)算法、Leaf-wise的決策樹生長策略、高效并行及Cache命中率優化等。
Histogram算法是對特征值進行裝箱處理,把連續的浮點型特征值離散化成k個整數,形成多個箱體(bin),同時構造一個寬為k的直方圖(如圖5所示)。在遍歷LUAD患者的IRG表達譜數據時先將浮點型的IRG表達值離散化,再把離散化后的值映射到相應的箱體中,當遍歷完一次數據后,對每一個箱體中的數據進行統計并繪制直方圖,然后根據直方圖的離散值,遍歷尋找最優分割點。這樣在訓練模型時就可直接使用離散化后的統計值,而不是原始的浮點型數據。這樣做的好處是減少特征的取值空間,從而提高模型的訓練效率和性能。此外,離散化后的特征也更便于進行特征的交叉和組合,以進一步提升模型的性能。值得注意的是,Histogram使用箱體代替原始數據,相當于增加了正則化,這意味著有更多的細節特征會被丟棄,相似的數據被分到同一個箱體中,所以箱體的數量決定了正則化程度,箱體越少懲罰越嚴重,過擬合的風險越低。
 圖5
Histogram算法裝箱處理過程
Figure5.
Histogram algorithm boxing process
圖5
Histogram算法裝箱處理過程
Figure5.
Histogram algorithm boxing process
GOSS算法通過對樣本采樣的方法來減少計算目標函數增益時的復雜度。在本實驗中,首先計算每個樣本的梯度并進行排序,然后選取梯度最大的a*100%個樣本作為集合A(實驗中a = 0.2),然后在剩下的數據中隨機采樣b*100%個樣本作為集合B(實驗中b = 0.3),之后計算信息增益時只在A∪B上運算,但為了保持數據分布不變,通常乘上系數(1 – a)/b來補償小梯度樣本。最后根據計算得到的信息增益對特征進行排序,以了解哪些IRG對預測結果有更大的影響。
EFB算法是處理現實生活中常見的高維數據、稀疏特征空間的方法(如圖6所示)。設特征1、特征2及特征3互為互斥的稀疏特征,通過EFB算法將三個特征捆綁為一個稠密的新特征,然后用該新特征替代原來的三個特征,從而實現不損失信息的情況下減少特征維度,提升梯度增強算法的速度。如本文中在使用LightGBM模型與XGBoost模型進行聯合分析篩選與LUAD預后相關的IRG時,就是將IRG表達譜數據隨機分組,然后對每一組數據進行分析,探尋IRG之間的相關性。如果當一個或幾個IRG表達值上升或下降時,其余的IRG表達值也隨之上升或下降,那么說明這些IRG之間具有相關性,此時選擇最具代表性的幾個IRG作為與LUAD預后相關的主要IRG特征進行保留。經過反復的實驗驗證后,對實驗結果取交集便初步篩選到了與LUAD預后相關的IRG。
 圖6
EFB算法特征處理過程
Figure6.
Feature processing process of EFB algorithm
圖6
EFB算法特征處理過程
Figure6.
Feature processing process of EFB algorithm
大部分決策樹的學習算法采用Level-wise策略生長樹,不加區分地一次分裂同一層的所有葉子,但很多葉子的分裂增益較低,所以該策略會產生許多不必要的分裂,消耗了更多的運行內存(如圖7所示)。LightGBM算法對它進行了改進,使用Leaf-wise策略生長樹,即每次只對分裂增益最大的葉子進行分裂,其他葉子不變。Leaf-wise策略可降低誤差,得到更好的精度,并能加快算法的學習速度(如圖8所示)。
 圖7
Level-wise策略生長樹
Figure7.
Level-wise strategy growth tree
圖7
Level-wise策略生長樹
Figure7.
Level-wise strategy growth tree
 圖8
Leaf-wise策略生長樹
Figure8.
Leaf-wise strategy growth tree
圖8
Leaf-wise策略生長樹
Figure8.
Leaf-wise strategy growth tree
總的來說,LightGBM是一個性能高度優化的GBDT算法,將LightGBM的優化用公式表達為LightGBM = XGBoost + Histogram + GOSS + EFB(如圖9所示)。其中,Histogram是直方圖算法,作用是減少候選分類點;GOSS是基于梯度的單邊采樣算法,作用是減少樣本數量;EFB是互斥特征捆綁算法,作用是減少特征數量。LightGBM支持高效并行訓練,且具有更快的訓練速度、更低的內存消耗、更好的準確率以及支持分布式快速處理海量數據等優點。
 圖9
LightGBM模型結構
Figure9.
LightGBM model structure
圖9
LightGBM模型結構
Figure9.
LightGBM model structure
將患者的K-means聚類結果和IRG表達譜數據納入LightGBM模型,用于預后模型的訓練和預測。在該項研究中,使用7/3拆分來生成訓練數據集和測試數據集,并選擇十折交叉驗證(cross-validation,CV)。
2 結果
本文使用XGBoost集成學習模型和BPNN深度學習模型作為基線模型。模型訓練完成后,使用受試者操作特征(receiver operating characteristic,ROC)曲線下面積(area under curve,AUC)來評估模型的效果(如圖10所示)。在測試集中,LightGBM模型的AUC值分別為0.96(第一組)、0.98(第二組)、0.96(第三組);在整個數據集中,LightGBM模型的AUC值分別為0.99(第一組)、0.99(第二組)、0.99(第三組)。在測試集中,對于第一組患者的生存率預測,XGBoost與LightGBM達到了相同的準確率(0.96),BPNN相對較差,僅為0.92;對于第二組和第三組患者的生存率預測,LightGBM模型預測效果優于XGBoost和BPNN模型。在整個數據集中,也只有在對第一組患者進行生存預測時,XGBoost模型能與LightGBM達到相同的準確率,而對于其他組患者的生存預測,LightGBM模型的效果優于XGBoost和BPNN模型。此外,通過LightGBM模型預測的患者表現出與通過K-means聚類的患者相似的生存結果,這也證實了LightGBM預測模型的準確性。本文提出的基于LightGBM的集成學習方法可將LUAD患者分為高生存率(5年生存率 ≥ 65%)、中等生存率(5年生存率 < 65%且 ≥ 30%)和低生存率(5年生存率 < 30%)三個組。
 圖10
模型性能的評估
Figure10.
Evaluation of model performance
圖10
模型性能的評估
Figure10.
Evaluation of model performance
3 討論
腫瘤異質性包括腫瘤間異質性(不同腫瘤細胞之間的基因與表現型不同)和腫瘤內異質性(相同腫瘤細胞內的基因與表現型不同),其中腫瘤內異質性又有空間異質性(相同腫瘤不同區域)和時間異質性(原發腫瘤和繼發腫瘤)之分[28]。因此,理想情況下,缺失值應替換為相同亞型腫瘤樣本中得出的平均值。由于數據來源是RNA-seq轉錄組測序,理論上,所有的基因都會被檢錄,絕大部分沒有被檢錄到的基因是因為它們的表達水平過低。所以,一般對轉錄組數據補缺失值的方法采用補0法,而我們采用的KNN方法是一種基于相似性的方法,它考慮了樣本之間的相關性,能更好地保留數據的整體結構。
準確預測癌癥患者的生存率對于治療決策十分重要。近年來,已有研究結果表明,IRG在LUAD的發生發展過程中發揮著重要的調控作用[29-31]。基于分子特征預測模型的開發為提高癌癥生存預測的準確性帶來了希望。但是,目前大多數基于分子的生存預測模型都是將患者分為兩組,其生存結果不同[32]。例如黃正品等[10]提出的LASSO-Cox模型和劉鳳燕等[33]提出的多因素Cox模型,將LUAD患者分為高低風險組,五年生存率預測(AUC值)分別為0.74和0.72。然而,這一結果對于治療決策來說不夠準確。在本研究中,使用LightGBM模型將LUAD患者分為三組,五年生存率預測AUC為0.96。
本研究從TCGA下載肺腺癌的RNA-seq數據和臨床數據,通過生物信息學方法和集成學習方法進行分析和挖掘,得到了17個IRG,其中KIF14、RHOV、S100P、AC025580.1、PCP4、TFAP2A、TNFSF11、ADRB2、AGER等IRG在早期的研究中已被提出[34-36]。本研究使用LightGBM模型新發現COL1A1、SIX1、FAM83A、PTPRH、RPL13AP17、WNT7A、GRIA1、GSTA3等8個IRG。預后分析結果顯示,這些IRG均與LUAD的預后相關(如圖3所示)。此外,有研究報道了COL1A1在NSCLC中表達高于正常組織[37],本研究的分析結果表明COL1A1與LUAD患者預后生存期呈負相關,更加驗證了COL1A1對LUAD的發生發展具有重要意義。
綜上所述,本研究分析新發現的8個IRG在LUAD組織與正常組織中的表達存在顯著差異,對LUAD預后評估有一定的參考價值,可作為LUAD的生物標志物進一步研究。因此,本研究的發現將有助于從分子機制了解LUAD的發生發展,并為LUAD的早期診斷、靶點治療和預后評估提供新的潛在生物學標志物。
4 結論
在這項研究中,我們基于TCGA數據庫和LightGBM集成學習方法構建LUAD患者的生存率預測模型,首次將LightGBM模型引入LUAD預后分析,成功克服了以往線性模型無法準確描述數據之間復雜非線性關系的局限,也克服了深度學習模型難以解釋的不足。通過利用非線性函數進行數據擬合,能夠更加精準地揭示數據之間的聯系,從而得到更精確的預測結果。該模型能夠將LUAD患者分為三組:5年生存率高于65%(第1組)、低于65%但高于30%(第2組)和低于30%(第3組),并且能較準確地預測LUAD患者的五年生存率。本模型的應用不僅提高了預測的準確性,而且使得結果更具可解釋性和可理解性,為醫生和患者提供了更可靠的參考依據。
但該模型也還存在一定的局限。首先,本研究從公共數據庫TCGA中獲取LUAD的相關數據,仍需要在多中心的大規模患者隊列中進一步驗證該模型的性能。此外,由于是基于生物信息學分析的研究,所以一些基因的預后性能需要進行相關的實驗來進一步證實。同時,雖然本研究是基于轉錄組測序數據進行的,理論上該模型具有應用于其他腫瘤預后評估的潛力,然而其實際表現尚未得到充分驗證,仍需進一步的研究。綜上所述,未來應該致力于驗證該模型在更大規模患者隊列中的表現,并通過實驗驗證相關基因的功能和作用機制,以進一步提高該模型的可靠性和準確性。另一方面還可以探索該模型在其他腫瘤預后評估中的應用潛力,為臨床實踐提供更多有效的工具。
重要聲明
利益沖突聲明:本文全體作者均聲明不存在利益沖突。
作者貢獻聲明:孟祥福主要負責總體解決方案設計、實驗指導、論文撰寫、論文審閱及修訂;田友發主要負責實驗方案設計、程序編寫、論文撰寫;張霄雁主要負責算法咨詢與建議。